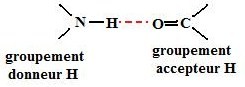Biochimie : étude de la matière vivante à l’échelle moléculaire.
Biotechnologie : application des principes scientifiques et de l’ingiéneurie à la transformation de matériaux par des agents biologiques pour produire des biens ou des services.
Introduction :
-acides aminés }
-peptides (moins de 100 AA) } composé azoté
-protéines }
Composition chimique : C, H, O, N et S (pour certain)
50 à 80% du poids sec
CHAPITRE 1 : LA STRUCTURE DES PROTEINES
1. Les acides aminés
1.1.Structure et classification des AA
1.1.1. structure générale :
COOH
/
H-C*-----------R
\
NH2
C* : carbone asymétrique ou chiral. Ce carbone confère à la molécule un pouvoir rotatoire.
Il existe 20 AA qui entrent dans la composition des protéines. La masse moléculaire est variable, on l‘estime en moyenne à 110 Da dans une chaîne poly peptidique.
- proline :} imine (amine secondaire) imuno-acide
- glycine :} pas de carbone asymétrique. C’est le plus petit AA.
1.1.2. Classification en fonction de la structure de R
- Chaîne aliphatique :
· Linéaire ou ramifié
· A fonction alcool
· A fonction souffré, thiol
· A fonction acide : -COOH
· A fonction amide : -CO-NH2
· A fonction basique
- Chaîne latéral cyclique
· Cycle aromatisé
· Hétérocycle non aromatisé
1.1.3. Classification en fonction de la polarité de R
Notion de polarité : C’est la capacité pour des molécules à établir des liaisons hydrogènes avec H2O ou avec un autre groupement polaire.
-AA à R polaire
R aliphatique ou cyclique sans groupement polaire. Ses AA sont qualifié d’hydrophobe. Plus la chaîne aliphatique est grande, plus ils sont hydrophobes.
-AA à R polaire, mais non chargé
-Ser+Thr+Tyr: AA hydroxylés
-Cys : thiol
-Asn, Gln : amide
-AA à R polaire chargé
Ils possèdent des groupements fonctionnels dont la charge en milieu aqueux va dépendre du pH.
R acide : Asp, Glu, (acide carboxyliques)
R basique : Lys, Arg, His
Renvoi : tableau 1 et 2 + figure 1
1.2.Stéréo-isomérie des AA
Pour chaque AA, sauf la glycine, il existe au moins un carbone chiral ; il y a donc 2 configuration possible suivant la position du NH2.
Représentation de Fischer (voir figure 2 stéréo-isoméries AA)
Ils y a au moins 2 énantiomères pour chaque AA, l’énantiomère D et l’énantiomère L.
Les AA naturels sont de série L.
Seul l’isoleucine et la thréonine ont 2 carbones asymétriques.
Racemisation : c’est le passage d’un énantiomère à un autre.
Elle s’accompagne le plus souvent de la rupture de la liaison covalente.
L’enzyme est la racemase, qui est produite par certaines bactéries.
2. Propriétés physiques des acides aminés
2.1.Solubilité dans l’eau
Ils sont tous soluble dans l’eau, avec des différences selon la nature de R.
· R ionisable : très soluble
· R non ionisable : soluble
· R apolaire : moins soluble
La solubilité dépend du pH : minimal au point isoélectrique (pHi)
pHi : c’est le pH auquel la charge net de la molécule est égale à 0, donc elle porte autant de charge positive que négative.
2.2.Solubilité dans les solvants organiques
Elle est plutôt faible en général. Elle est aussi différente suivant le R des AA.
On l’utilise pour identifier les AA, par la séparation sur solvant, de la méthode de la chromatographie.
2.3.Pouvoir rotatoire
C’est la capacité pour une solution d’AA à dévier le plan de polarisation d’une lumière polarisé d’un angle alpha
Si α supérieur à 0, la solution est alors dextrogyre.
Si α inférieur à 0, la solution est lévogyre.
Voire la loi de Biot/cour de physique
Cette propriété est dût à la présence d’au moins un carbone asymétrique.
2.4.Absorption des rayonnements UV
Aucun AA absorbent la lumière visible.
Certains absorbent la lumière UV : ce sont tous des AA aromatiques.
Ce sont les doubles liaisons conjuguées des cycles aromatiques qui sont responsable de l’absorption.
Les absorptions à une longueur d’onde inférieure à 250 nm sont non spécifiques car de nombreuses molécules absorbent à ces mêmes longueurs d’onde.
Chromophore : c’est une molécule qui absorbe la lumière (visible ou UV)
2.5.Fluorescence
C’est une longueur d’onde émise supérieur à la longueur d’onde excitatrice.
Les molécules qui émettent de la fluorescence sont des fluorophores. On peut faire un dosage par fluorimetrie.
On peut surtout doser la tyrosine et le tryptophane.
3. Propriétés chimiques des AA
3.1.Propriété acido-basique
Ils ont au moins deux fonctions ionisable, la fonction acide carboxylique et la fonction amine.
3.1.1. dissociation des AA à R neutre
-Dissociation de –COOH
COOH+H2OèCOO-+H3O+
pKa d’environ 2
-Dissociation de NH2
NH3++H2OèNH2+H3O+
-Diagramme de distribution des espèces en fonction du pH.
-Courbe de titrage (cf. Fig. 5)
-pHi= (pK1+pK2)/2
-Pour chaque équilibre de dissociation :
pH<pKa [acide]>[base conjuguée]
pH>pKa [acide]<[base conjuguée]
-Zone de pKaèzone de tampon _ demi dissociation.
3.1.2. Dissociation des AA à R. acide
Asp, Glu
-Nombre de groupements ionisable : 3
-ordre de dissociation : pK1èpKRèpK2
-pHi= (pK1+pKR)/2
(Entre les 2 dissociations acides)
3.1.3. Dissociation des AA à R. basique
His, Lys, Arg
-Nombre de groupements ionisable : 3.
-Ordre de dissociation : pK1èpK2èpKR pour la lysine et l’arginine.
pK1èpKRèpK2 pour l’histidine.
-pHi= (pKR+pK2)/2
(Entre les 2 pKa des fonctions basiques)
Ampholyte=amphotère : molécule qui se comporte comme un acide ou une base selon le pH.
-Les AA ont un pouvoir tampon si pH=pKa
-Les AA en solution dan l’eau pure : pHsol=pHi (forme zwitterion)
3.2.Autres propriétés chimiques des AA
-Propriétés dues à la fonction acide carboxylique (peu réactif, fonction stable)
-Propriétés dues à la fonction amine :
Basique (capte un H+) etc… NH2 : groupement très réactif à cause du doublet non liant du N.
-Propriétés dues à la chaîne latérale.
4. Importance biologique de certains AA
Les AA standards : protéinogènes (20 AA)
- rôle structural : leur enchaînement détermine la structure et la fonction des protéines. Il y a 7 AA essentiels : l’organisme ne peut pas les synthétiser. Ce sont la leucine, l’isoleucine, la lysine, la méthionine, la phénylalanine, la thréonine et le tryptophane.
- Rôle énergétique : ils peuvent être utilisés comme source d’énergie comme les glucides et les lipides.
Les AA non standards : ce sont des AA modifiés, non incorporés dans les protéines. Ex. : histamine (dérivé de l’histidine). Ils ont des rôles physiologiques variables.
Les AA et la biotechnologie : la lysine est un AA essentiel qui est présent en très petite quantité dans les protéines végétales. La nourriture des herbivores doit souvent être supplémenté en lysine, produit par les IAA (Industrie AgroAlimentaire).
Technique de production è génie fermentaire : culture en fermenteur de Corynbacterie naturellement productrice de lysine.
5. Les polypeptides
5.1.La liaison peptidique
5.1.1. définition
C’est une liaison covalente qui s’établit par condensation entre le groupement carboxylique d’un AA et le groupement amine du suivant.
5.1.2. Propriétés de la liaison peptidique
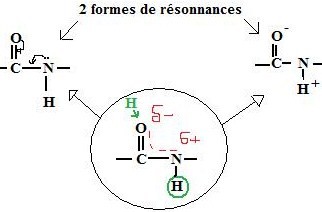
Etude au rayons X : mesure de la liaison peptidique.
Elle est plus courte que la longueur théorique.
Conclusion : on n’a pas un vrai liaison simpleèphénomène de résonnance.
Conséquence : lumière polariséeèpossibilité d’établir des liaisons H
èStructure tridimensionnelle de la protéine
La liaison peptidique est plane car elle a le caractère d’une liaison double partielle. Les 6 atomes de sont bloqués dans le même plan. C’est le plan peptidique. Il est rigide. Il n’y a pas de rotation possible entre le C et l’azote.
Le plan peptidique est bloqué en configuration trans (voir fig. 3)
èRotations des plans peptidiques (fig. 4)
Au niveau du Cα, il existe deux possibilités de rotations définis par les angles ψ et ω. Les différentes valeurs de ces angles de rotations des plans peptidiques déterminent la structure secondaire de la chaîne peptidique. Mais toutes les configurations ne sont pas permises (voir fig. 5).
èConclusion
Le polypeptide est un enchaînement linéaire (non ramifié) et flexible de petit plan correspondant au liaisons peptidiques. Chaque plans est rattachés à ses voisins au niveau d’un pivot : le Cα. Les rotations des plans assurent le replis de la chaîne et permettent l’établissement d’une structure secondaire soit répétitive (si ψ et φ sont identique à chaque fois) soit non répétitive (si φ et ψ sont différents à chaque fois)
5.1.1.1.Caractéristique des chaînes peptidiques
1) vectorisées (sensèordre des AA)
2) Les AA engagés dans la chaîne sont des résidus, car ils ont perdu un molécule d’eau. Masse moyenne d’un résidu : 110 Da. Ils portent le suffixe –yl à la place de –ine.
3) Les AA N. terminales sont représentés à gauche (NH2 de libre)
Les AA C. terminales sont représentés à droite (COOH de libre)
4) La numérotation des AA se fait de gauche à droite (de NèC)
2 AAè dipeptide
3 AA è tripeptide….
N ≤ 10 = oligopeptide
N > 10 = polypeptide
5.1.1.2.Détermination de la séquence primaire de la chaîne peptidique
Structure primaire : correspond à l’ordre d’enchaînement des AA par liaison peptidique depuis N. terminale à C. terminale.
Il existe de nombreuses combinaisons possibles pour une séquence de N AA pris au hasard parmi les 20. Pour une protéine de N AA, il existe 20N.
L’ordre est déterminé génétiquement.
5.1.1.2.1. Analyse de la composition globale en AA.
Hydrolyse des liaisons peptidiques

Inconvénient, le tryptophane est détruit, il faut donc réaliser un dosage UV avant l’hydrolyse.
L’asparagine et la glutamine ont leur fonction amide libérée (NH3).
On va obtenir la formule brut du peptide : Ala, Ser, Gly3…
5.1.1.2.2. Identification des extrémités N et C terminales
A l’aide de réactifs chimiques
- Réactifs d’Edman : PTC è on obtient un PTH-AA (N. terminale)
- Réactif de Sanger : DNFB è on obtient BNP-AA (N. terminale)
- Chlorure de dansyle (N. terminale)
A l’aide de peptidases è exopeptidases
- carboxypeptidases
- amidopeptidases
A partir de ces peptides, on peut faire une cinétique (suivre l’ordre des AA terminaux)
5.1.1.2.3. Fragmentation de la chaîne peptidique : endopeptidase (fig. 2)
5.1.1.2.4. Séquençage des fragments (fig. 5)
Se fait par la méthode récurrente d’Edman. Cette méthode et la méthode de référence : elle permet d’enlever un part un les résidus en commencent par les extrémités N. terminales. Elle nécessite que l’extrémité NH2 soit libre.
L’analyse consiste en des cycles répétitifs d’action du réactif suivit de l’analyse du PTH-AA libéré à la fin de chaque cycle par HPLC.
Chaque cycle dure une heure. La méthode est aujourd’hui automatisée : un séquenceur automatique et couplé à une HPLC permet de séquencer des fragments d’environ 20 résidus (fig. 7)
5.1.1.2.5. Nouvelles coupures et agencement des fragments séquencés (fig. 6)
5.1.1.2.6. Localisation des ponts dissulfures

Ils sont en intra ou interchaine.
Nouvelle électrophorèse : apparition d’une nouvelle tache (localisation du pont dissulfure)
5.1.1.2.7. Séquençage par spectrométrie de masse
Le peptide est fragmenté et bombardé par un gaz rare, ce qui produit des molécules chargées identifiées par leur différence de masse. Ceci nécessite un traitement informatique des données obtenues. Cette technique est complexe, coûteuse, mais de plus en plus utilisé (car rapide, séquence de moins de 30 résidus)
5.1.1.2.8. Bilan
Le premier peptide séquencé fut l’insuline en 1953, par Sanger (prix Nobel), 51 résidus.
Aujourd’hui, la méthode est automatisée « micro-sequencage » selon la méthode d’Edman.
Le séquençage est une étape importante dans l’étude des protéines. Les séquences obtenues sont répertoriées dans les banques de données (voir cour protéines). Cependant, la majorité des séquences protéiques répertoriées sont issu du séquençage des gènes. C’est à partir des gènes que l’on déduit la séquence des protéines, à l’aide du code génétique.
5.1.1.3.Détermination de la masse moléculaire d’un peptide
5.1.1.3.1. Par électrophorèse : SDS.PAGE
Il s’agit de faire la migration du peptide dans un champ électrique en présence de marqueur de taille.
5.1.1.3.2. Par chromatographie è gel filtration (exclusion diffusion)
5.1.1.3.3. Par calcul à partir du poids de la séquence
Estimation : Masse molaire moyenne d’un résidu : 110 Da
M estimation = 110 * n résidus
Réel : somme des masses molaires de chaque résidu.
5.1.1.4.Détermination du pHi
5.1.1.4.1. Définition
C’est le pH pour lequel la charge globale de la molécule est nulle : autant de charge positives que négatives.
Le pHi est fonction des pKA des chaînes latérales et des extrémités terminales. 85% des protéines ont un pHi entre 4.5 et 8.5.
5.1.1.4.2. Méthodes expérimentales
- Courbe de titrage
- Isoélectrofocalisation : c’est une technique d’électrophorèse.
5.1.1.4.3. Par calcul : renvoi cours
5.1.1.5.Structure tridimensionnelle des protéines
5.1.1.5.1. Les différents niveaux de structure
- structure primaire : définit par les liaisons covalentes entre AA.
Se détermine par le séquençage des protéines isolées et purifiées.
Se détermine par le séquençage du gène codant pour la protéine.
- structure secondaire : liaison hydrogène entre les AA proches dans la chaîne peptidique.
- structure tertiaire : différente liaison entre AA éloignés dans la chaîne peptidique.
-structure quaternaire : association des différentes sous unité des protéines multimériques (plusieurs chaînes peptidiques). La structure quaternaire n’existe pas chez toutes les protéines.
5.1.1.5.2. Les structures secondaires
5.1.1.5.2.1.Etude théorique
Les plans peptidiques ne peuvent pivoter qu’autour des carbones asymétriques, avec les angles ψ et φ. Seul certaines valeurs peuvent être prises, et correspondent aux structures suivantes :
Ce sont des structures ordonnées et répétitives.
Il existe aussi des coudes et des pelotes statistiques, qui ont des structures ordonnées non répétitives. Ses structures ont été confirmées par l’analyse de cristaux de protéines aux rayons X.
5.1.1.5.2.2.L’hélice α
Liaisons stabilisantes : liaison entre l’hydrogène et l’oxygène de la liaison peptidique impliquant le résidu N et l’azote et l’hydrogène de la liaison peptidique impliquant de résidu N+4.

Autres hélices : il existe aussi des hélices :
· De proline
· 310
· Gauche (collagène)
5.1.1.5.2.3.Le feuillet β
AA formant un feuillet β : tryptophane, phénylalanine, tyrosine.
Structure répétitive ordonnée, plissée, étirée (non enroulée).
- Les liaisons stabilisatrices sont de nature hydrogène dans la liaison peptidique, impliquant des résidus se retrouvant en face à face dans l’espace, mais relativement proche dans la structure primaire.
- Caractéristique géométrique :
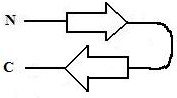
Selon l’orientation des chaînes, on observe :
· Des feuillets parallèles
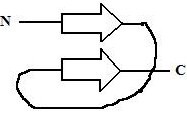
· Des feuillets antiparallèles
5.1.1.5.2.4.Tours, coude et boucle (β turn)
Ce sont des structures secondaires non répétitives, constituées de 2 à 6 AA. Stabilisé par des liaison hydrogène entre CO et NH, et permettent de connecter les hélices et/ou les feuillets.
5.1.1.5.2.5.Structure en pelote statistique
Ce sont des structures ordonnées non répétitives, très flexibles qui vont pouvoir s’adapter à la structure complémentaire d’une autre molécule.
Ex : - se trouve en pelote statistique le domaine hypervariable des Ig.
- le site actif des enzymes è reconnaît S.
5.1.1.5.2.6.Les superstructures secondaires : les motifs
Ce sont des assemblages type de structure secondaire qui se rencontre au sein des structures tertiaires et qui correspondent à la succession de certaines structures secondaires.
En doigt de zinc : sont caractéristiques des protéines qui se fixent sur l’ADN. Facteur de transcription.
5.1.1.6.1. La structure tertiaire des protéines.
Elles consistent en l’organisation des structures secondaires entre elles. Ce n’est pas une structure figée, elle peut se modifier sous l’effet de la fixation d’une autre molécule ou sous l’effet de variation du pH ou de la température. C’est la structure tertiaire qui confère ses propriétés biologiques à la protéine.
5.1.1.6.1.1. Les liaisons stabilisatrices faibles
Ce sont des liaisons qui s’établissent entre les chaînes latérales des résidus éloignés dans la séquence.
- Les liaisons hydrogène : partagent d’un hydrogène entre 2 atomes électronégatifs.
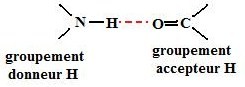
- Les liaisons ioniques : qui vont s’établir entre 2 groupements de charge contraire.
Ex. : COO--NH3+. Cette liaison peut être altérée par un traitement au NaCl.
- Interaction entre dipôle
- Interaction hydrophobe : ce sont des forces d’interaction entre des molécules non polaire permettant de minimiser leur contact avec l’eau.
5.1.1.6.1.2. Les liaisons fortes ; covalente (ponts dissulfure entre les cystéines)
Ces ponts dissulfure s’établissent après l’acquisition de la structure 3D. Toutes les cystéines ne forment pas de ponts dissulfures.
5.1.1.6.1.3. Notion de domaine
C’est une partie de la chaîne peptidique qui peut se replier indépendamment du reste de la chaîne pour former un domaine fonctionnel à activité biologique précise.
Ex. : Domaine variable et domaine constant des immunoglobulines (Ig).
5.2.Structure quaternaire des protéines
Elle ne concerne que l’association des différentes sous unités d’une protéine : protéine oligomérique.
Ex. : l’hémoglobine est une protéine tétramérique, composée de 2 sous unités α et de 2 sous unités β.
Les sous unités peuvent être identiques ou différentes.
Les liaisons stabilisatrices sont de nature faible (liaison hydrogène, ionique…).
Intérêt de l’association : permet le contrôle de l’activité biologique de la protéine.
Ex. : La protéine kinase A possède 4 sous unités catalytiques. C’est seulement lorsque les sous unités sont dissociés qu’elle a une activité catalytique.
6. Les méthodes d’analyse de la structure tridimensionnelle des protéines
6.1.Approche de la bioinformatique : analyse prédictive à partir de la séquence primaire
Soit à l’aide de logiciel : il nous donne la probabilité d’avoir une hélice α, un feuillet β…, connaissant la séquence en AA.
Par comparaison dans les PCB « Protein Data Bank ». Il recherche les homologies entre la protéine inconnue, appelé X, et des protéines dont la structure est connue.
6.2.Approche biophysique
6.2.1. La cristallographie
Après obtention d’un cristal (à partir de la protéine, qui a été extraite et purifié), celui-ci est soumis aux rayons X : ceux-ci sont diffractés par le cristal puis analysés pour donner ne carte de densité électronique permettant de reconstituer la répartition des atomes dans la protéine.
6.2.2. RMN
C’est une technique basée sur le comportement des atomes, soumis à un champ magnétique.
7. Dénaturation renaturation des protéines
7.1.Définition
Dénaturation : perte de la structure tridimensionnelle par rupture des liaisons à faible énergie et/ou réductions des ponts dissulfures. Il y a atteinte des structures quaternaire, tertiaire et secondaire, sans hydrolyse des liaisons peptidiques, donc sans atteinte sur la structure primaire.
7.2.Les étapes
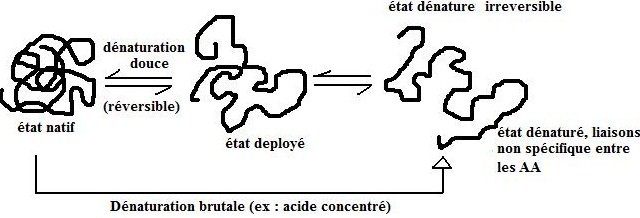
7.3.Les agents dénaturants
7.3.1. Les agents physiques
· La température supérieur à 45°C : rupture des liaisons hydrogène (thermo coagulation de l’albumine du blanc d’œuf)
Remarque : Les micro-organisme thermophiles : leurs protéines sont plus résistantes à la température (elles sont riches en pont dissulfure)
· Les ultrasons (toutes les liaisons sont affectées)
· Les ultraviolets (toutes les liaisons sont affectées)
· Variation brutale du pH è réaction irréversible
7.3.2. Les agents chimiques
· Les acides et les métaux lourds è irréversibles (précipitation, coagulation…)
· Les milieux fortement concentrés en sels, à force ionique élevée : ils ont un effet de relargage. Les sels captent les molécules d’eau qui, normalement, participent à la solubilité des protéines. Le manque d’eau force les protéines à exposer leurs AA hydrophobes en surface, permettant ainsi les contacts hydrophobes entre protéines, qui agglutinent et précipitent. On peut utiliser la précipitation sélective pour séparer différentes protéines (voir cour de seconde année). C’est un phénomène réversible, donc très utilisé.
· L’urée : agent chaotropique, qui participe à la rupture des liaisons hydrogènes. (Réversible)
· β mercaptoéthanol : agent réducteur des ponts dissulfures. (Réversible)
· SDS : c’est un détergeant anionique
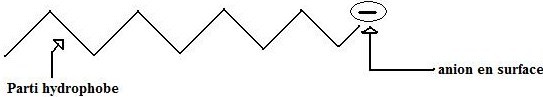
7.4.Conséquences
Une protéine dénaturée va perdre :
· ses propriétés biologiques (protéine active uniquement sous une forme tridimensionnelle donnée)
· sa solubilité è précipitation
Les protéines sont des molécules fragiles, il faut travailler à 4°C (cela évite l’action des protéases), en milieu tamponné, à force ionique moyenne (eau physiologique à 9 pour mille) avec un agent protecteur.
8. Maturation et adressage des protéines
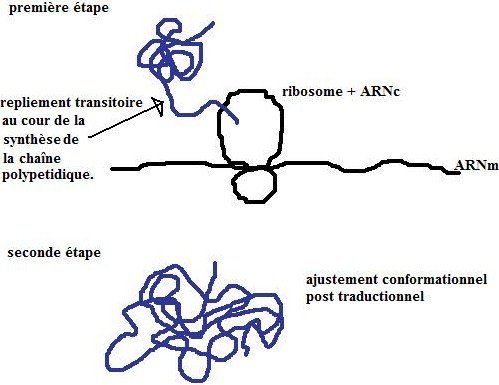
8.1.Repliement des protéines : folding
Objectifs :
· Acquérir une structure particulière reconnue par un ligand
· Enfouir les AA hydrophobes
8.1.1. Définition
C’est le processus par lequel la chaîne polypeptidique linéaire d’une protéine acquiert une structure tridimensionnelle. Ceci va lui permettre de reconnaître spécifiquement un ligand. Des milliers de protéines doivent reconnaître chacune un ligand qui lui est propre, les structures des protéines doivent donc être très diverses et variées.
8.1.2. Mécanisme
Le mécanisme reste encore inconnu. L’objectif de la protéine est la réduction de son entropie (encombrement), pour que celle-ci dépense le moins d’énergie.
La durée du repliement est de l’ordre du millième de seconde. (10^-3 seconde)
On obtient une structure native unique.
8.1.3. Expérience de ANFINSEN (Nobel 1972)
C’est une expérience de dénaturation renaturation. Il a montré que après la renaturation de la protéine, celle-ci retrouve la même structure tri dimensionnelle initiale. Dans un environnement approprié, toute l’information nécessaire au repliement de la protéine est contenue dans sa séquence primaire.
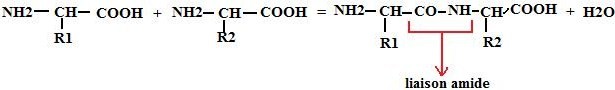

 Ils sont en intra ou interchaine.
Ils sont en intra ou interchaine.