Les corps de Cajal (CB)
(issu de la Review The Cajal Bodies, de Glenn E. Morris, 2008, Biochimica et Biophysica Acta)

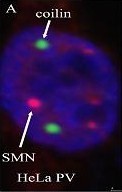
La protéine marqueur, la p80 coïline, a été identifié comme auto-anticorps humain, et les anticorps contre cette protéine ont depuis été exclusivement utilisé pour définir les CBs dans les cellules de mammifère. De plus, la coïline apparait être essentiel à l’intégrité et à la fonction des CBs. Les CBs sans coïline, aussi connu comme « résidu de CB », ne reste quand la coïline est enlevé expérimentalement, mais sont capable de recruter les snRNPs d’épissage. De plus, la coïline des CBs n’apparait pas être essentiel pour l’épissage car les souris KO coïline -/- peuvent survivre, même si elles ont une viabilité réduite. On peut donc en déduire que les CBs augmentent l’assemblage des snRNPs en concentrant les enzymes et les substrats en une région du nucléoplasme : en absence de CBs, les mêmes réactions ont lieu avec une plus faible efficacité dans le nucléoplasme.
Les CBs sont des structures dynamiques. En premier lieu, ils n’existent pas dans tous les types de cellules, même si les cellules HeLa « rapide » ne sont que 5 à 10% à manquer de CBs. De plus, certains types cellulaires manquent de CBs visibles (comme les cellules de peau). Il a été suggéré que l’abondance des CBs est déterminée par le niveau d’expression des snRNPs et de la quantité d’ARN présent dans le noyau, même in vivo. Le nombre de CBs peut donc varier rapidement s’il y a un changement du niveau de transcription.
Les CBs peuvent se déplacer librement dans le nucléoplasme. Le photobleaching suggère un échange rapide et continu des protéines contenues dans le CB et le nucléoplasme. Même si la coïline et SMN ont un temps de rétention de quelque minute, et que les snRNPs ont un temps de rétention beaucoup plus court, de l’ordre des 30 secondes. Cela est cohérent dans le sens que la coïline et SMN se trouve aussi en dehors des CBs, dans le nucléoplasme ou dans le cytoplasme.
L’épissage, rappel :
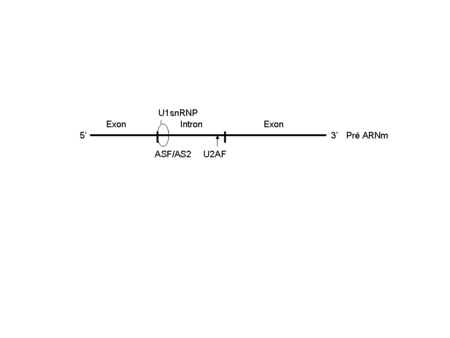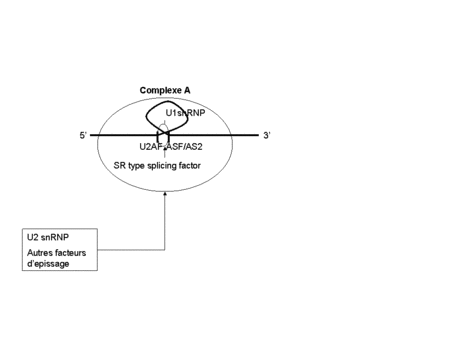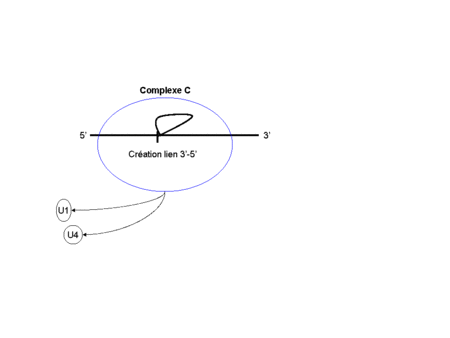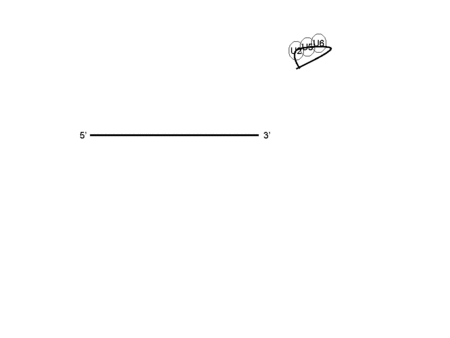 Des 7 snRNP, seul 5 font partit du spliceosome (splice = épissage) Les 5 snRNPs varient énormément au niveau de leur structure et de leur complexité.
Des 7 snRNP, seul 5 font partit du spliceosome (splice = épissage) Les 5 snRNPs varient énormément au niveau de leur structure et de leur complexité.
Pour assembler le spliceosome, l’U1 snRNP s'attache à sa cible, au niveau de la séquence 5' de chaque intron du preARN, aidé par un facteur de transcription, ASF/AS2. La séquence en 3' de l'intron est reconnue par le complexe U2AF (U2 snRNP auxiliary factor). D'autres facteurs d'épissage de type SR forment un pont entre les 2 sites, pour former le complexe E.
Ce complexe est capable de lier U2 snRNP et d'autres facteurs de transcription pour former le complexe A, ou "pre-spliceosome".
Le "tri SNP" est formé dans le Corps de Cajal, formé d’U4, U5 et U6 snRNP. L'addition du Tri snRNP au spliceosome forme le complexe B (requière le facteur de transcription SPF30, protéine présentant une homologie avec SMN).
Un réarrangement des snRNPs, après la perte d’U1 et U4, lie les sites d'épissage en 5' et 3' dans le C complexe, et l'intron "lariat" est excisé avec les U2, U5 et U6 snRNP encore attaché.
Plus de 150 protéines sont nécessaires pour cette dernière étape, et l'épissage alternatif est contrôlé par les protéines facteurs SR. Cette complexité est encore augmentée par le fait qu'une minorité de préARN sont épissés par un système alternatif où U1, U2, U4 et U6 snRNP sont remplacés par U11, U12, 4atac et 6atac snRNP.
Formation des snRNPs
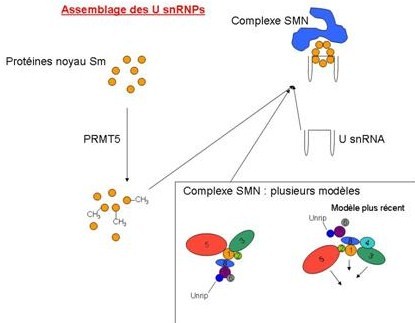
Les U snRNPs sont formés d’U snRNAs plus 7 protéines noyau Sm (Smith), qui forment un anneau. Cette assemblage est possible grâce à un complexe de 9 protéines, le complexe SMN. La structure et l’organisation du complexe SMN émerge lentement. Les protéines du complexe sont appelées gemin, et ont chacune une fonction précise :
- gemin 1, ou SMN, est capable de se lier au snRNA
- gemin
- le sous complexe gemin3/4 peut aussi avoir une fonction indépendante de SMN, comme être un composant majeur des microRNP (miRNP)
- gemin 3 est prédit comme une boite DEAD de
- Gemin 6 et 7 ont une taille et une similitude structurale par apport aux protéines Sm, suggérant que ces gemins ont pour acte d’assembler les protéines noyaux Sm sur le U snRNA (il n’y a pas d’évidence que ce tremplin/raccourci soit nécessaire)
- Gemin 5 est un membre de la famille des WD-repeat dans l’assemblage des protéines, et lie le snRNA au complexe SMN dans le cytoplasme en reconnaissant des séquences spécifiques.
- Unrip est une autre protéine appartenant au WD-repeat, mais ne semble pas requise après l’assemblage des snRNPs par le complexe SMN dans le cytoplasme. Il a également été suggéré qu’unrip puisse également être impliqué dans la phosphorylation cytoplasmique de SMN, qui est par la suite inversé par une étape de déphosphorylation dans le noyau.
Il reste encore à découvrir beaucoup de chose sur SMN, comme la réaction permettant aux U snRNPs de se détacher, ou encore la composition et le comportement de SMN après son action.
Une fois dans le CB, le complexe SMN voit se détacher gemin5. Pour que le complexe SMN cible les CBs, il faut une interaction avec la coïline, rendu possible par une association fermé entre la coïline et SMN dans les CBs. Pour détacher le snRNP, la coïline entre en compétition avec les domaines RG des protéines Sm, lié à SMN.
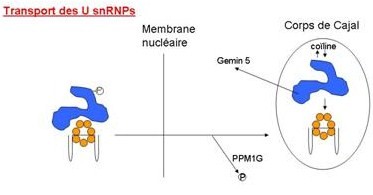
Les thermes corps de Cajal peut être utilisé quand la coïline et SMN sont présents (situation normal), le corps de Cajal sans SMN quand seul la coïline est présente, et gem quand SMN est seul.
La viabilité (cependant réduite) des souris KO coïline -/- sans CB met en évidence le fiat que la fonction des CB est d’augmenter l’efficacité de processus de formation des U snRNPs. De plus, sous certaines conditions apparait le fait que seule une fraction de la coïline, snRNP ou du complexe SMN est localisé dans les CBs. En contraste avec les souris coïline -/-, les souris KO SMN-/- présentent une mortalité embryonnaire, concordant sur le fait que SMN joue un rôle essentiel dans les modifications et l’assemblage des snRNPs.
Plus loin encore, les CBs permettent l’assemblage du U2 snRNP, inclus l’addition des facteurs d’épissage SF3a et SF3b en complexe oligomérique et l’assemblage du U5 snRNP avec U4/U6 après chaque cycle d’épissage pour reformer le « tri snRNP ».
D’autre modifications sont possibles comme :
- une 2’-O-ribose methylation
- une pseudouridylation
Les CBs permettent aussi :
- la modification des snRNA, s’il y a présence des scaRNA (smal DB RNA) qui guident les enzymes de modification à la séquence cible et sont analogues au scoRNA (smal nucleolar RNA) impliqué dans l’orientation des enzymes pour la modification de la bonne séquence d’ARN ribosomique dans le nucléole
- 
- les CBs sont aussi impliqué dans le processus de réplication dépendant des ARNm des histones. Ces derniers n’ont pas d’introns, donc ne subissent pas l’épissage, mais U7 snRNP se fixe à leur extrémité
- les CBs peuvent également s’associer avec les gènes d’U2 snRNP d’une manière complètement différente qu’avec l’association des gènes histoniques. Le lien est la transcription dépendante dans ce cas et l’association des CBs avec les nouveaux transcrits ARN depuis le gène U2 snRNP, même si la raison pour laquelle ils s’associent avec les gènes U2 snRNP avant, comme après, à l’assemblage des U2snRNP dans le cytoplasme n’est pas encore clair. Lorsque le CB est lié au locus U2 snRNA, il est souvent lié au corps PML, qui est riches en facteurs de transcription, et ce lien est consolidé par une interaction entre la coïline et la protéine PML, PIASy
- chez Xenopus, l’ARN polymérase 2 pourrait être préassemblé dans le CB, parce que le complexe SMN interagirait avec le domaine C terminal (l’hélicase A) et les mutants dominants négatifs de SMN inhibe la polymérase 2 et cause son agglutination dans les structures contenant la coïline.
En conclusion :
Les CBs ont pour rôle principaux l’assemblage final des U snRNPs, ainsi que diverses fonctions se réalisant sous certaines conditions (peu connu), dont la modification des U snRNA.
Il faut cependant insister sur le fait qu’ils ne sont pas indispensables : les réactions peuvent se réaliser dans le nucléoplasme, même si l’effet est apparemment moins important.
Comme vous avez put le voir, les recherches sur le CB se concentrent surtout sur le complexe SMN, qui, lorsqu’il est muté, provoque une maladie héréditaire connu sous le nom de SMA, soit l’atrophie musculaire spinale (Spinal Muscular Atrophie).
Pour en savoir plus sur cette maladie : ici