Organisation du génome humain
1.La double hélice d’ADN
Rappel : constituée de 2 chaines de polydesoxyribonucléotides, antiparallèles.
-
4 bases azotées : 2 puriques : adénine A et guanine G
2 pyrimidiques : cytosine C et thymine T
Les liaisons hydrogènes entre les bases azotées sont complémentaires :
G et C : 3 liaisons ; A et T : 2 liaisons
Les bras forment une double hélice avec un pas de 3,4 nm
-
2 types de sillon : 1 petit et 1 grand. Le grand interagit avec les protéines de transcription.
-
2 formes d’ADN : l’ADN B, tourne à droite (cas général)
L’ADN Z, riche en G-C, hélice gauche (en zigzag)
La séquence d’ADN contient le code génétique avec 64 codons de 3 nucléotides. Un codon correspond à un acide aminé. Le code génétique est universel et dégénéré. Il y a un codon d’initiation à la transduction, un pour la traduction et 3 codons stop (UGA, UAA et UAG)
2.Compactage de l’ADN dans le noyau
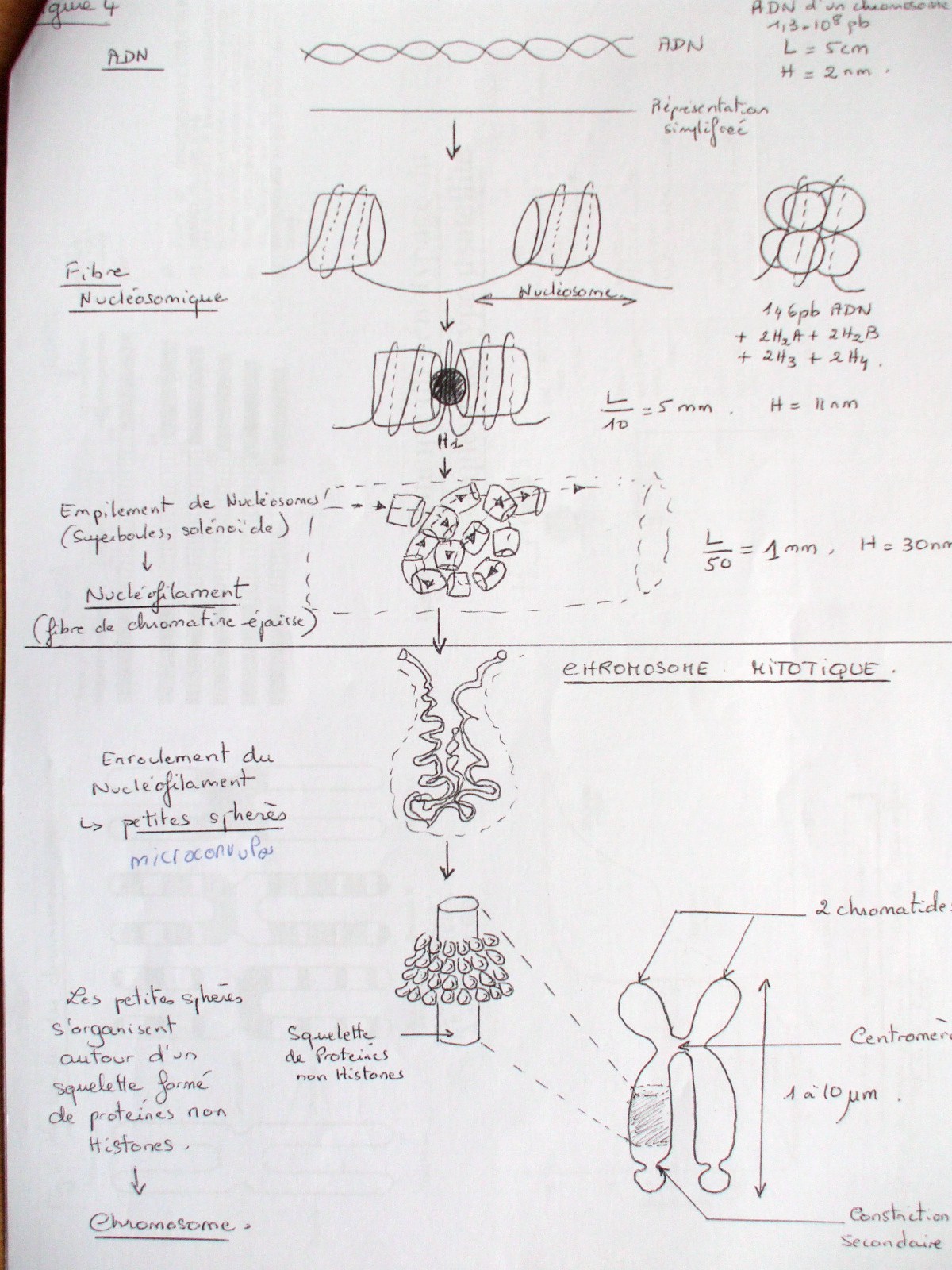
L’ADN génomique humain fait 3.000.000.000 pb et 1 mètre de longueur, qui doivent entrer dans le noyau. Il y a donc besoin d’un compactage énorme.
Il y a d’abord des fibres de 100 Ä (0,1 nm) de diamètre constituées de nucléosomes (146 pb + 8 histones). H1 ressert les nucléosomes entre eux. Les histones peuvent être phosphorylés, acéthylées, methylées et ADP ribosylées : ces modifications post-traductionnelle permettent un décompactage de la chromatine nécessaire à la réplication de l’ADN et sa transcription.
Il y a ensuite un solénoïde de 300 Ä de diamètre appelé nucléofilament, stabilisé par les HMG (High Mobility Group) pour donner la chromatine.
Il existe deux catégories de chromatine :
L’euchromatine, qui est une chromatine décondensé qui correspond à une fraction active des gènes pour la transcription bande q et g sur le chromosome
L’hétérochromatine qui est une chromatine très compacté en général au niveau des centromères et qui correspond à la fraction inactive des gènes pour la transcription (=région riche en C-G) bande C
Pendant la mitose et la méiose, la chromatine se compact encore pour donner des chromosomes. L’ADN forme des petites sphères appelées microconvules qui se rattachent sur un squelette de Scaffold Protein. Un chromosome possède 2 chromatides reliées par un centromère.
- Metacentrique
- Acrocentrique
- Telocentrique
L’Homme possède 22 paires de chromosomes homologues (1 exemplaire du père et 1 exemplaire de la mère) plus une paire de chromosome sexuelle.
Caryotype : ensemble des chromosomes
Pour se repérer sur les chromosomes et les distinguer entre eux, on effectue différentes colorations :
-
Coloration à la quinacrine : bande Q visible par fluorescence
-
Protéases + Giemsa : bande G
Les bandes Q et G se superposent et correspondent en général à des régions plus riches en A-T.
-
Dénaturation + acridine orange : cela donne des bandes R complémentaires aux régions G et Q. C’est donc une région riche en G-C.
-
Dénaturation/renaturation : bandes C au niveau des centromères, qui est de l’hétérochromatine toujours compacté.
3.Les différents types de séquences dans l’ADN
Exp : Courbe de renaturation de l’ADNg humain
Cette courbe montre qu’il existe 3 catégories d’ADN différents, correspondant aux 3 sigmoïdes successives ayant des Cot1/2 différents (Concentration en ADN fois le temps de demi-dénaturation)
-
ADN hautement répétitif : réappariement quasiment instantané
-
ADN moyennement répétitif : réappariement plus lent
-
ADN à séquence unique : renaturation très lente
3.1.ADN hautement répétitif
3.1.1.ADN « satellite » (environ 15% de l’ADNg)
-
Séquence CEN (centromérique) appelé aussi « α satellite) Motif de 171 paires de base répété n fois, riche en G-C, dans les centromères. C’est l’ADN correspondant aux bandes C. Cette région servirait d’ancrage aux protéines du kinetochore pour fixer le chromosome aux microtubules du faisceau mitotique.
-
Séquence TEL (télomérique) : le motif TTAGGG est répété des milliers de fois. Le motif est rajouté à l’extrémité des chromosomes par la télomérase pour protéger les télomères vis-à-vis des nucléases.
3.1.2.Mini-satellite ou VNTR (Variable Number of Tandem Repeat)
Ce sont des séquences de 11 à 16 pb répétées en tandem avec un nombre de copie variable d’un individu à l’autre. Il peut y avoir jusqu’à 1000 répétitions de suite. Les séquences sont dispersées dans tout le génome (surtout dans les télomères) excepté sur le chromosome X ou Y. Ils sont très polymorphes car polyallèlique (jusqu’à 1000 allèles différents par VNTR)
N.B. : PIC : Polymorphism Information Content
Plus le PIC est élevé (1), plus les séquences sont hyper informatives pour distinguer 2 individus.
3.1.3.Microsatellite ou STR (Short Tandem Repetition)
Ce sont des motifs très court (de 1 à 4 pb) répété en tandem jusqu’à 40 fois. Ils sont très abondants (environ 130.000). Ils sont répartis de facon uniforme sur tous les chromosomes, et sont très polymorphes comme les VNTR. Ils proviennent sans doute d’un dérapage de l’enzyme de réplication.
Les VNTR et STR sont analysés par PCR en utilisant des amorces dans les régions flanquantes de ces satellites. On obtient des amplicons qui diffèrent les uns des autres par peu de paire de bases (2 par exemple), et sont donc séparés sur gel de polyacrilamide.
3.2.ADN moyennement répétitif
Il représente 25% du génome
3.2.1.ADN non codant
Séquences qui sont des retrotransposants (élément génétique mobile et retro : utilisation de la transcription inverse)
SINE : Short Interspersed Repetitive Element.
Ex : séquence Alu : séquences de 300 pb, avec 900.000 copies/génome humain
Ces séquences présentent une homologie avec l’ARN 7SL (cet ARN appartient aux particules qui interagissent avec le peptide signal des protéines à exporter à travers la membrane)
LINE : Long Interspersed Repetitive Element
Ex : line 1, 6 à 7 kpb, environ 5.000 à 10.000 copies /génome humain
Possède : ORF1 (OpenReading Frame) : protéine de 38 kDa, rôle inconnu
ORF2 : code pour une transcriptase inverse
3.2.2.ADN codant
-
Les gènes ribosomiques (ADNr vers ARNr). Ces gènes sont regroupés en Clusters de 200 copies d’une unité de transcription, et ceci sur 5 chromosomes différents. L’ARN polymérase 1 donne un ARN unique de 45S qui donnera ensuite 3 ARNr de 28S, 18S et 5,8S. Ces Clusters d’ADNr correspondent aux constrictions secondaires des chromosomes et aux nucléoles dans la chromatine inter phasique.
-
Les gènes pour ARNt. Ils sont aussi regroupés en Clusters avec des répétitions en tandem. Il y a environ 1.200 copies d’ADNt par génome humain. Ils sont transcris par l’ADN polymérase 3.
3.3.Séquences uniques codant pour des protéines
Dans le génome, il y a environ 30.000 gènes, ce qui représente seulement 5% du génome.
3.3.1.Structure d’un gène
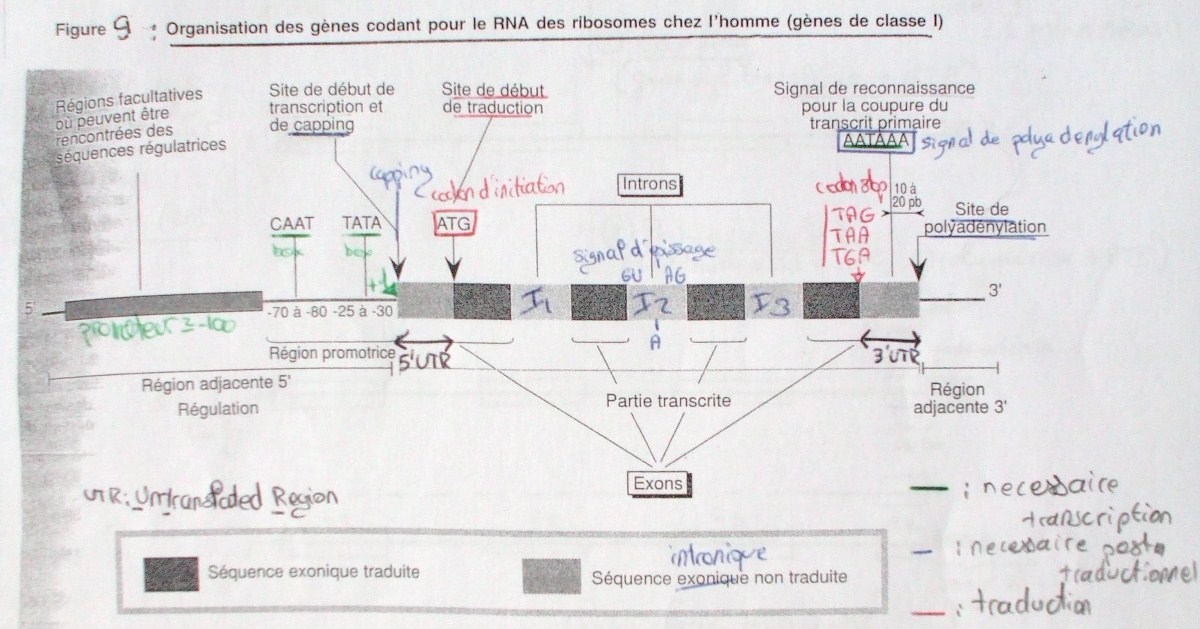
Les signaux nécessaires à la transcription
-
Le promoteur (-100 pb), en amont du gène : c’est le site d’arrimage des transrégulateurs (transactivateurs et transreppresseurs) qui régulent l’efficacité d’initiation de la transcription.
-
CATT box (-70 à -80)
-
TATA box (-25 à -30), existe aussi chez les bactéries.
-
Site +1 : site de démarrage de la transcription.
-
Site AATAAA : site d’arrêt de la transcription.
Les signaux de modifications post-traductionnelles
-
Site de capping en 5’ (ajout de la coiffe)
-
Signal de polyadenylation en 3’ (ajout d’une queue polyA 20 nucléotides après)
-
Signaux d’épissage dans les introns, GU en 5’, AG en 3’ et le site de branchement A une vingtaine de nucléotides avant AG.
Les signaux nécessaires à la traduction
-
Codon d’initiation à la traduction
-
Codon stop
3.3.2.Les 2 catégories de gènes
Les gènes de ménage (house kaping genes) : ce sont des gènes essentielles à la survie cellulaire et qui sont donc exprimé dans toutes les cellules de l’organisme (=ubiquitaire)
Ex : enzyme de réparation de l’ADN, protéine ribosomiale, enzyme de la glycolyse…
Les gènes tissu-spécifique : ce sont des gènes exprimés seulement dans certains tissus et qui sont à l’origine de la différenciation cellulaire.
Ex : insuline dans les cellules β des îlots de Langerhans du pancréas endocrine, kératine dans les cheveux, ongles…
3.3.3.Famille de gène : famille multigénique
C’est un regroupement de plusieurs gènes homologues au niveau d’un même locus génétique. Ces familles sont issues de la duplication d’un gène ancestral suivit d’une divergence par mutation.
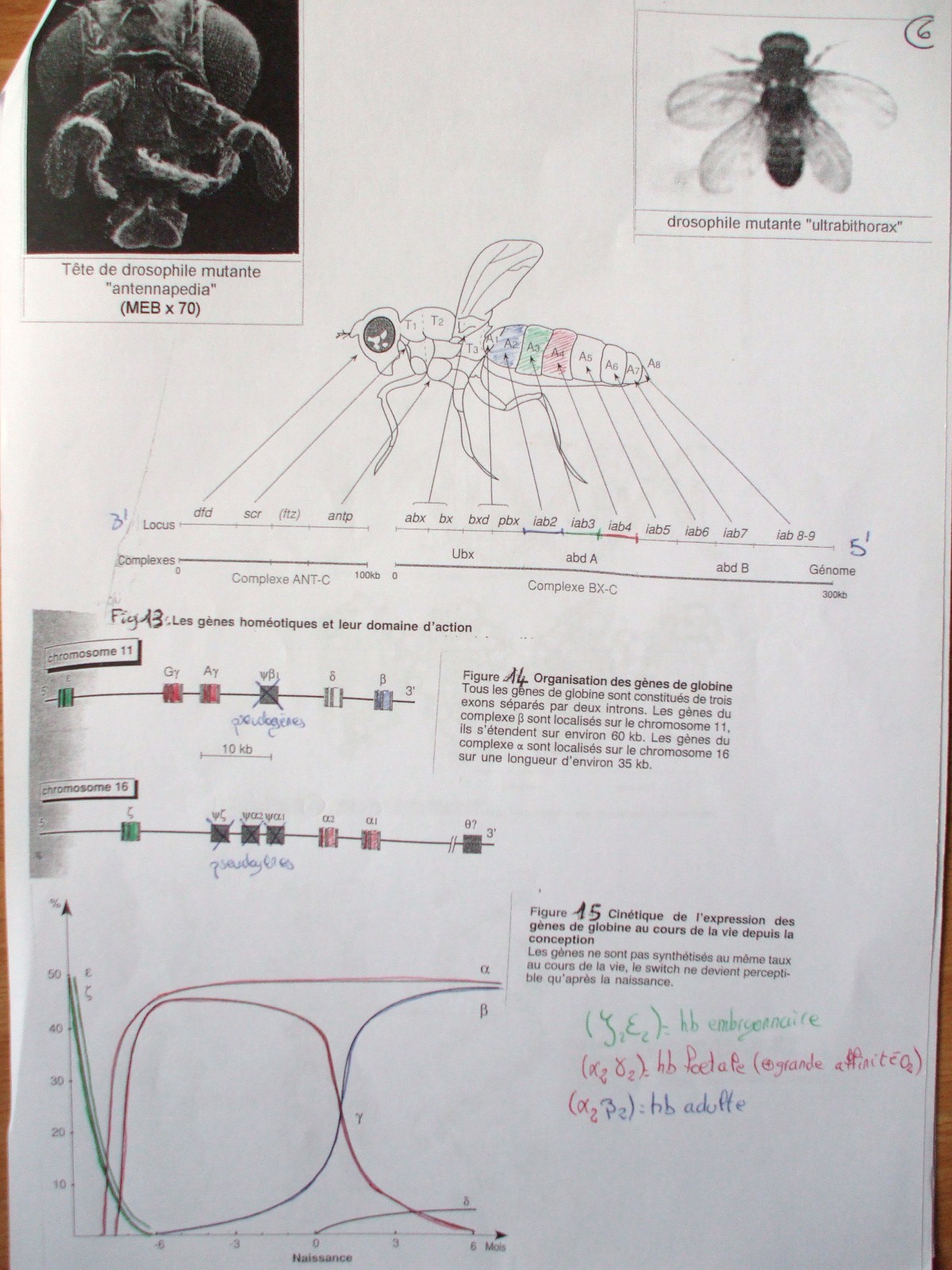
Ex1 : famille des homéogènes : (en bas)
Ces gènes sont responsables de la différenciation des segments chez la drosophile. Les gènes ont une organisation très particulière car leur ordre 3’=>5’ sur l’ADN correspond à un ordre d’expression entero-posterieur dans le corps de la drosophile. Chaque segment n+1 exprime un gène de plus que le segment n => Colinéarité spatiale
Ex 2 : famille des globines chez l’Homme (en bas)
Il existe 2 loci pour la globine. Le locus α sur le chromosome 16 et le locus β sur le chromosome 11. On constate cette fois-ci une colinéarité temporelle, c'est-à-dire une correspondance entre l’ordre des gènes 3’=>5’ sur l’ADN et leur expression au cours de l’ontogénèse (formation du corps)
3.3.4.Pseudogène (φ)
Ce sont des copies non fonctionnelles d’un gène ni transcris, ni traduit. Les pseudogènes peuvent provenir de 2 mécanismes de duplication
-
Incomplète, le gène est tronqué
-
Complète, mais ensuite suivi de mutation (sur le promoteur, le codon d’initiation, stop…)