Le noyau, un organite intracellulaire hautement organisé
Le noyau est un organite cellulaire hautement structuré dans lequel ont lieu deux fonctions essentielles à la cellule : la réplication de l’ADN et la transcription des gènes cellulaires. On sait maintenant que l’expression des gènes cellulaires est soumise à plusieurs niveaux de régulation : le premier provient de la séquence d’ADN elle-même, notamment des séquences promotrices. Ainsi, la présence de motifs reconnus par des facteurs de transcription dans les promoteurs des gènes cellulaire détermine leur expression au niveau basal dans divers tissus ainsi que leur induction ou répression en fonction de divers stimuli externes. Le second niveau de régulation est épi-génétique : l’ADN est enroulé autour d’histones dont le niveau de compaction et les modifications post-traductionnelles (acétylation, méthylation, ubiquitinylation) déterminent l’expression d’un gène. On a récemment découvert que l’expression d’un gène est aussi régulée par son positionnement dans le noyau. En particulier, il apparait que la transcription d’un gène donné peut être régulée par sa position à l’intérieur du noyau et par son association avec des domaines nucléaires. Cette dernière notion implique l’existence de domaines nucléaires qui ont des fonctions qui leur sont propres et qui les différencient les uns des autres. En effet, le noyau est hautement organisé, et la plupart des événements nucléaires n’ont pas lieu dans tout le nucléoplasme, mais sont limités à des sites spatialement restreints (97).Avant la découverte des domaines nucléaires, il y a un peu plus de deux siècles, F. Fontana remarqua la présence d’un corps rond dans le noyau : le nucléole (54). C’est le premier corps nucléaire identifié, ainsi qu’un des plus importants. C’est avec l’évolution des techniques que le rôle du nucléole fut, petit à petit, compris. Ainsi, la présence d’ARN fut mise en évidence à l’intérieur de ce corps dans les années 1950, et l’identification des gènes ribosomaux (ADNr) dans la région nucléolaire organisée a été possible dans les années 1960 (113). Il a alors été proposé que la transcription des gènes ribosomaux et la biogénèse des ribosomes aient lieu au sein du nucléole. L’organisation fonctionnelle du nucléole a aussi été explorée, en grande partie grâce à l’amélioration des techniques de microscopie électronique. A l’aide d’une analyse au spectromètre de masse de composants extraits de nucléoles purifiés, il a récemment été mis en évidence que le nucléole n’est pas uniquement le lieu de biogénèse des ribosomes. En effet, cette analyse a identifié environ 700 protéines nucléolaires, dont plusieurs n’ont aucun rôle dans la biogénèse des ribosomes. Cette observation permet d’avancer que le nucléole est un corps nucléaire multifonctionnel (5).
Les premières suggestions montrant que le noyau cellulaire dispose de territoires chromosomiques a été faite en 1885 par Rabl, qui a observé dans les noyaux de cellules épithéliales de Salamandra des territoires, et a émis l’hypothèse qu’un territoire chromosomique était construit d’une chaine de chromatine primaire, à partir duquel se forment des structures secondaires et tertiaires, formant ainsi la chromatine (117). En 1909, T. Boveri émit les principales hypothèses concernant ces domaines chromosomiques, la première étant les territoires chromosomiques sont maintenus de façon stable durant l’interphase et la seconde que l’arrangement des territoires chromosomiques est conservée entre la cellule mère et les deux cellules filles (revue en (31)). Cependant, l’apparition de la microscopie électronique au milieu des années 1950 ne révèle pas la présence de domaines chromosomiques individuels, et l’existence même de ces territoires est alors remise en cause. Ainsi, la plupart des chercheurs dans les années 1970-80 étaient convaincus que le noyau était rempli d’un entremêlement et de boucles de chromatine comme dans une assiette de spaghetti. C’est à partir de 1977 que l’équipe de S. Stack démontra par plusieurs expériences que les chromosomes étaient contenus dans un espace nucléaire précis (revue en (31)). Depuis il a été montré à de nombreuses reprises que la chromatine est hautement organisée, et les différents territoires chromosomique humains ont été définis par des expériences d’hybridation in situ (FISH) en 2005 (14). Malgré tout, la chromatine d’un chromosome n’est pas cloitrée dans son territoire chromosomique, mais comporte des boucles qui peuvent s’étendre en dehors du territoire chromosomique auquel elles appartiennent. De plus, les chromosomes ne sont pas répartis aléatoirement dans le noyau de la cellule. Ainsi, les chromosomes pauvres en gènes ont tendance à se positionner vers l’extérieur du noyau, tandis que les chromosomes riches en gènes vont se placer à l’intérieur. Cette disposition est conservée à travers les espèces et est déterminée par plusieurs facteurs : la densité locale en gènes, l’activité transcriptionnelle, la chronologie de réplication et la quantité de séquences riches en GC. Cependant, ceci est une généralisation car certaines cellules comme les fibroblastes ont une distribution des chromosomes en fonction de leur taille et donc indépendamment de la densité de gènes.
Après la confirmation de l’existence des territoires chromosomiques, il a été mis en évidence que ces territoires sont séparés par le compartiment inter-chromatinien (3). Le compartiment inter-chromatinien commence au niveau des pores nucléaires et s’étend dans de larges canaux et lagunes entre les territoires chromosomiques. Il pourrait représenter le lieu où se produit la réplication de l’ADN (73), et même des événements de réparation de l’ADN (136). De plus, il se trouve dans cet espace une grande variété de corps nucléaires, tels que les corps de Cajal, les corps PML, les « speckles » (ou domaine SC35), les paraspeckles, les corps PolyComb (PcG) et les foyers de transcription (138). La structure et les fonctions de ces corps nucléaires sont décrites en détail dans les paragraphes suivants.
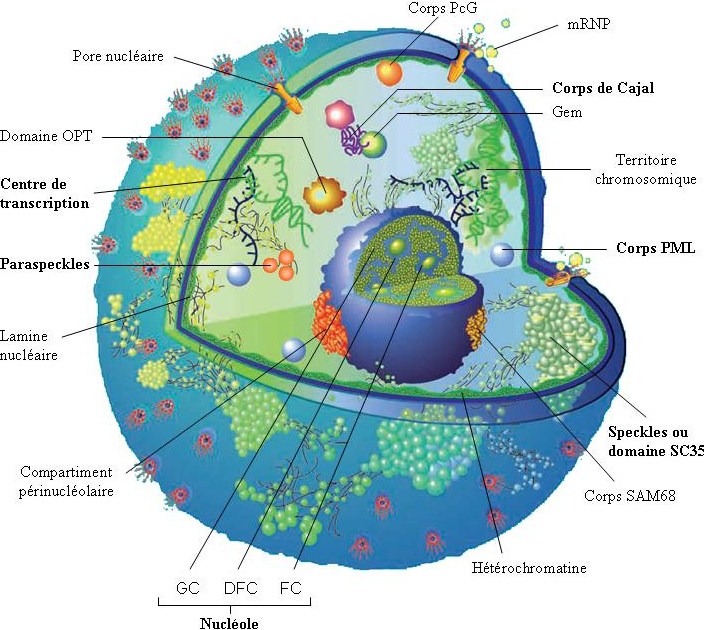
Figure : Organisation des domaines et corps nucléaires. Représentation schématique
tridimensionnelle de l’organisation d’un noyau de cellule eucaryote. Celui-ci est composé de
différentes zones et corps nucléaires, dont le nucléole, les domaines SC35, les corps PML, les
corps de Cajal, les foyers de transcription et les paraspeckles, qui seront décris en détail plus
bas (adapté de (138)).
1 Les corps nucléaires, présentation et biogénèse
Comme souligné précédemment, les domaines ou corps nucléaires identifiés dans le noyau ne sont pas séparés du nucléoplasme par une membrane lipidique. Ces domaines nucléaires sont définis par l’accumulation d’un certain nombre de protéines et éventuellement d’acides nucléiques. A l’exception du nucléole qui est visible en microscopie optique, les autres domaines sont généralement identifiés à l’aide de techniques d’immunofluorescence en utilisant des anticorps reconnaissant les constituants typiques du domaine, par exemple la protéine PML, constituant le corps du même nom, ou encore la protéine SC35 dans le cas des domaines SC35. Ces corps compartimentent le volume nucléaire pour créer des sites distincts dans lesquels sont concentrés des réactifs afin de faciliter des réactions biologiques. Récemment, plusieurs études se sont intéressées à la composition de ces domaines et aux mécanismes responsables de l’assemblage et de la maintenance de plusieurs corps nucléaires (5, 75, 90, 130).Il y a une grande diversité dans la taille et le nombre de corps nucléaires par noyau. Ainsi, le nucléole atteint une taille de plusieurs micromètres, tandis que les corps de Cajal, dont le diamètre est au maximum de 1,5 µm peuvent être au nombre de dix par noyau. La taille, l’aspect et le nombre de corps nucléaires dépend généralement du type cellulaire, et peut aussi changer en fonction de stimuli externes ou de l’état de la cellule, en particulier sa transformation en cellule cancéreuse. La plupart des corps nucléaires sont présents tout au long de l’interphase, et durant ce temps ils bougent dans le noyau, et ces mouvements sont généralement inversement proportionnels à leur taille (43). Ainsi, les domaines SC35 qui ont une taille de plusieurs micromètres sont généralement immobiles ou retenus à un espace local (78), tandis que les corps PML ou de Cajal (environ 1 µm) effectuent de plus grands mouvements et présentent de nombreux événements de fission ou de fusion (43).
L’étude des corps nucléaire a montré que les protéines qui les composent sont aussi présentes à des concentrations plus faible dans le nucléoplasme avec lequel ont lieu des échanges fréquent (42). Afin d’observer la dynamique des corps nucléaires, il a été réalisé des expériences de photo-blanchiment. Cette technique permet, en détruisant la fluorescence d’une protéine fusion en un point précis de la cellule, de mesurer le recouvrement ou l’extinction de la protéine fusion fluorescente au cours du temps dans la zone ciblée. Ceci permet ainsi de mesurer le temps de demi-recouvrement de fluorescence, qui dans le cas des corps nucléaire est très rapide. Ces résultats indiquent donc que la plupart des composants des corps nucléaires présentent un taux d’échange rapide entre le corps et le nucléoplasme (40, 114). L’apparence d’un corps nucléaire est ainsi largement déterminée par les interactions entre ses composants. Pour le moment, aucune interaction covalente entre protéines n’a été identifiée comme déterminante dans la structure des corps nucléaires, et il semble que la plupart des interactions dans ces corps soient faibles et non-covalentes.
La formation de certains corps nucléaires a été étudiée, et trois modèles sont proposés pour expliquer leur assemblage (41, 90). Selon le premier modèle d’assemblage stochastique, le processus d’assemblage des composants du corps nucléaire est aléatoire et ne respecte pas d’ordre hiérarchique. Chaque composant intervient de façon équivalente dans l’assemblage de ce corps nucléaire. Le second modèle prévoit, à l’opposé, un mode d’assemblage ordonné. Ce modèle propose que les étapes d’assemblage soient étroitement contrôlées par l’ordre de présentation des composants du corps. Il y aurait ainsi un ordre hiérarchique des composants, ce qui fait que seule une ou un nombre limité de voies pourraient permettre l’assemblage de ce type de corps nucléaire. Un troisième modèle intermédiaire est l’assemblage en « graine », selon lequel le corps nucléaire résulte de l’assemblage ordonné d’un nombre limité de composants, qui attirent ensuite les autres composants de façon aléatoire.
Les prochains paragraphes vont se focaliser sur la description d’un certain nombre de corps nucléaires, leur formation et leur maintien dans le noyau de la cellule.
1.1 Le nucléole
Le nucléole est un corps nucléaire extrêmement complexe et très étudié et seules ses fonctions et interactions principales seront décrites ici.Le nucléole est un domaine intranucléaire jouant un rôle très important dans la synthèse des ARN ribosomique (ARNr) et l’assemblage des ribosomes. Les gènes ribosomaux (ADNr) sont présents en multiples copies dans le nucléole. Chaque unité d’ADNr consiste en une séquence transcrite et une région non transcrite dans laquelle se trouvent toutes les séquences nécessaires pour la transcription par l’ARN polymérase I, comme les promoteurs proximaux, les promoteurs distants et les terminateurs. C’est à partir de ces ADNr que sont transcrits, dans le nucléole, les ARNr permettant la synthèse des ribosomes.
Le nucléole est composé de différents compartiments, observables par microscopie électronique (ME). On peut distinguer trois compartiments nucléolaires principaux : le centre fibrillaire (FC), le composant fibrillaire dense (DFC) et le composant granulaire (GC). Le FC est une région claire en ME, partiellement ou totalement entourée par le DFC, tandis que le GC occupe le reste de l’espace. Suivant le type cellulaire, le nucléole présente un nombre variable de FC de différentes tailles, avec une proportion inverse entre la taille et le nombre. Généralement, les cellules ayant un haut taux de biogénèse des ribosomes possèdent plusieurs petits FC. Les ARNr naissants apparaissent à la jonction entre FC et DFC, puis s’accumulent dans le DFC (67). Le GC est le lieu où différentes étapes de la maturation des pré-ribosomes s’effectuent. Lorsque la biogénèse des ribosomes est active, le confinement de certaines machineries dans les FC, DFC ou GC rend possible le marquage de ces compartiments par immunofluorescence.
De récentes analyses des composants du nucléole ont mis en évidence qu’il n’est pas uniquement composé de protéines impliquées dans la biogénèse des ribosomes. On peut ainsi trouver des protéines de régulation du cycle cellulaire comme CDK2, la protéine p53, des protéines chaperonnes ou des protéines impliquées dans l’épissage d’ARNm (5). Tandis que les signaux qui gouvernent la localisation nucléaire et l’exportation nucléaire des protéines sont maintenant bien définis, ceux concernant leur localisation dans le nucléole sont encore mal compris. Ainsi, plusieurs protéines localisées dans le nucléole contiennent une séquence de localisation nucléolaire (NoLS) qui est probablement impliquée dans la rétention nucléolaire en permettant l’interaction avec une molécule nucléolaire (par exemple B23). Cependant, la présence d’un NoLS dans la séquence d’une protéine n’est pas suffisante pour prédire sa localisation dans le nucléole. Il a récemment été identifié sur des protéines une séquence de rétention nucléolaire capturée par des ARNnc au niveau du nucléole, et on pourrait ainsi penser que la rétention nucléolaire soit un mécanisme post-traductionnel de la protéine permettant sa rétention et/ou son relargage du nucléole (8). Il a également été montré que les protéines nucléolaires s’associent et se dissocient rapidement des autres composants nucléolaires, permettant des échanges continus entre le nucléole et le nucléoplasme (114).
De très nombreuses études ont étudié la relation entre transformation cancéreuse et activité du nucléole. En effet, il est possible d’évaluer le potentiel de prolifération d’un cancer en mesurant le taux de biogénèse des ARNr du nucléole des cellules tumorales. De plus, de nombreux proto-oncogènes et suppresseurs de tumeurs s’y trouvent localisés et affectent la production de ribosomes. Il a aussi été mis en évidence que la diminution de la production de ribosomes induit l’apoptose, tandis que la perturbation du nucléole stabilise p53 (revue en (121)).
1.2 Les corps PML
Les corps PML sont des domaines sphériques d’un diamètre compris entre 0.1 et 1µm et qui peuvent avoir un centre micro-granuleux. Leur nombre est généralement compris entre 5 à 20 corps par noyau, et ce sont des corps protéiques ne contenant pas d’ADN ou d’ARN. Ils ont été visualisés pour la première fois par microscopie électronique dans les années 1960 (34), puis par immunofluorescence à l’aide d’un sérum auto-immun reconnaissant la protéine SP100, un des composants de ces corps nucléaires. Plus tard, les protéines PML (Promyelocytic leukemia protein) ont été identifiées comme étant un des constituants majeurs de ces corps, et essentielles à leur assemblage. Ces protéines appartiennent à la famille protéique TRIM/RBCC, dont beaucoup des membres sont des ubiquitine ligases. La transcription du gène PML est étroitement contrôlée par les interférons α/β et γ, mais aussi par la protéine p53 (33), chaque facteur pouvant augmenter de façon drastique la taille et le nombre des corps PML. La protéine PML subit plusieurs modifications post-traductionnelles, dont des phosphorylations et des sumoylations. La sumoylation de PML est impliquée dans la biogénèse de ces corps nucléaires (voir plus loin). De plus, les dommages à l’ADN ou les kinases activées par le stress phosphorylent PML, et peuvent ainsi réguler la stabilité de PML, la biogénèse de ces corps nucléaires et leurs associations et finalement contribuer à la réparation de l’ADN ou le contrôle de l’apoptose.Comme les autres corps nucléaires, les corps PML présentent un haut taux d’échange protéique entre le corps et le nucléoplasme. Cependant, dans le cas des corps PML, on remarque que les protéines partenaires bougent plus rapidement que les protéines PML. Les corps en eux même ne sont pas extrêmement mobiles, même si des événements de fission et de fusion peuvent être observés (102).
Une des propriétés les plus intéressantes des corps PML est la sumoylation. SUMO est une petite protéine semblable à l’ubiquitine, qui est conjuguée de façon covalente aux protéines cibles sur des résidus lysine, créant une branche de peptides et modifiant leurs propriétés.SUMO est impliqué dans de multiples voies, généralement comme un régulateur des interactions protéiques. De plus, SUMO peut interagir avec un court motif, appelé motif d’interaction avec SUMO (SIM). PML contient un SIM ainsi que trois sites de sumoylation, et l’interaction entre PML-SUMO et SIM est proposé comme étant responsable de la biogénèse de ces corps nucléaires (93).
L’étude de la biogénèse de ces corps a été grandement facilitée par l’utilisation de trioxyde d’arsenic. Le trioxyde d’arsenic est le seul composé qui permet, d’une manière séquentielle, la formation de ces corps nucléaires, la sumoylation de PML puis le recrutement des partenaires (82). Cependant, le mode d’action même du trioxyde d’arsenic n’est pas encore totalement compris. Il a de plus été mis en évidence que plusieurs partenaires des corps PML possèdent le domaine SIM, mais que leurs sumoylation n’est pas obligatoire (27).
La fonction des corps PML est encore mal connue, mais deux informations se dégagent de leur étude : ils pourraient d’abord servir comme un site de dépôt de protéines cellulaires, qui pourraient alors être facilement mises à disposition par relargage à partir de ces sites. C’est le cas de la protéine DAXX dont la séquestration au sein des corps PML permettrait de réguler l’apoptose. La deuxième fonction pourrait être la modification post-traductionnelle des partenaires de ces corps, dont le plus étudié est p53. Il a aussi été rapporté que les corps PML peuvent dégrader leurs partenaires, car ils concentrent aussi protéasome et ubiquitine (82).
1.3 Les domaines SC35
Dans les cellules de mammifère, les constituants de la machinerie d’épissage des ARN pré-messager (préARNm), incluant les snRNP (small nuclear Ribonucleoprotein), certaines sous-unités du splicéosome et d’autres facteurs d’épissage non snRNP, montrent une distribution caractéristique en tache ou speckles. Chaque speckle constitue un domaine nucléaire nommé également domaine SC35, du nom d’un de ces constituants protéiques majeurs, la protéine SC35 (150). Ces corps nucléaires, initialement décrit par Ramon y Cajal en 1910 (revue (80)), ont été visualisés par microscopie électronique en 1959 par Swift (143) qui les a nommés granules inter-chromatinien (ICG). Il est maintenant clair que les facteurs d’épissage se trouvent dans les domaines SC35 ou ICG, qui ont une taille comprise entre un et plusieurs micromètres de diamètre et sont composés de granules de 20 à 25 nm reliés par une mince fibrille, donnant ainsi une apparence en collier de perles.Les domaines SC35 se situent dans l’espace inter-chromatinien, dans des régions où ne se trouvent que peu ou pas d’ADN. Ils sont cependant souvent associés à des gènes transcriptionnellement actifs (17), même si cela ne semble pas obligatoire pour leur transcription ou épissage. Ainsi, il a été trouvé que les régions chromosomique riches en gènes (bandes R) sont localisés plus près des domaines SC35 que les régions pauvres en gènes (bandes G) (131). De plus, les gènes ayant une activité coordonnée (gènes de même famille par exemple) peuvent se retrouver associés à un même domaine SC35, même si ces gènes se trouvent sur des chromosomes différents.
Plusieurs publications mettent en évidence que les domaines SC35 agissent comme des sites d’assemblage, de stockage et de modification des facteurs d’épissage des pré-ARNm. Il a aussi été montré que les pré-ARNm naissants sont localisés préférentiellement à l’extérieur des domaines SC35 dans des structures fibrillaires de 3 à 5 nm de diamètre, lesquelles sont appelés fibrilles péri-chromatiniennes, tandis que les ARNm épissés sont localisés à l’intérieur du domaine SC35 (28).
La composition des domaines SC35 a été identifiée par l’analyse protéomique de fractions d’ICG enrichies. Cette approche a identifié 146 protéines, dont plusieurs sont à ce jour non caractérisées (123). Parmi les protéines connues, la plupart ont un lien avec la transcription et l’épissage des ARNm. On trouve ainsi dans les domaines SC35 des facteurs de transcription, plusieurs sous unités de l’ARN polymérase II, des facteurs de prise en charge des extrémités 3’ des ARN et des protéines d’épissage. De plus, une population d’ARN poly(A)+ est localisée dans les domaines SC35. Parmi ces ARN poly(A)+, Malat1 a été identifié comme étant un long ARN non codant enrichi dans les domaines SC35. Il a été montré que Malat1 interagit avec le domaine de liaison à l’ARN des protéines d’épissage SR (10), et joue un rôle dans la régulation de l’épissage alternatif en modulant la phosphorylation du facteur ASF/SF2, un des constituants des domaines SC35.
Un article récent a identifié la protéine Son comme nécessaire à la constitution des domaines SC35 (128). Son est une protéine concentrée dans les domaines SC35, et possède en plus d’un domaine RS riche en arginine-sérine, de multiple répétitions de sérines réparties tout du long de sa séquence, lui permettant de se lier à de nombreux partenaires. Ces interactions font de Son un composant essentiel dans l’organisation des domaines SC35, aussi bien lors de l’interphase que de la mitose. Des études supplémentaires sont cependant nécessaires afin de mieux caractériser ses fonctions.
Comme d’autre corps nucléaires, les domaines SC35 sont des structures dynamiques, leur taille, forme et nombre varient en fonction du type cellulaire, suivant le niveau d’expression des gènes et en réponse aux signaux pouvant influencer la transcription et/ou l’épissage. En effet, l’augmentation de l’expression de gènes contenant des introns, entraine une diminution de l’accumulation des facteurs de transcription et/ou d’épissage au sein des domaines SC35, et leur redistribution vers des sites de transcription actifs. Les composants individuels des domaines SC35 peuvent ainsi faire la navette entre les sites de transcription et ce corps nucléaire. Afin de visualiser les mouvements de ces composants, des expériences de photo-blanchiment ont été réalisées en utilisant la protéine ASF/SF2 fusionnée à la GFP. Il a ainsi été montré que la durée de demi-recouvrement de la fluorescence est de 3 à 5 secondes, avec un recouvrement total d’environ 30 secondes, démontrant ainsi un mouvement d’échange très rapide de ce composant (114).
L’étude des domaines SC35 a servi de modèle pour comprendre la maintenance des corps nucléaires. Plusieurs études ont mis en évidence que les domaines SC35 se forment probablement par auto-assemblage suggérant le modèle stochastique, et que leur morphologie est déterminé par le niveau d’interaction entre les macromolécules (revue en (98)). Des ARNnc sont aussi présents dans les domaines SC35, et ils peuvent avoir un effet sur le recrutement de protéines SR ainsi que sur l’expression de certain gènes (10). A l’état d’équilibre, les taux d’association et de dissociation des composants des domaines SC35 vont définir la taille du corps et la quantité de composants présents dans le nucléoplasme. Des mécanismes de régulation peuvent influencer les taux d’association et/ou de dissociation, et ainsi changer les quantités de la fraction fixée et soluble en réponse à des signaux cellulaires spécifiques. Au final, la taille et l’aspect des domaines SC35 résultent de la dynamique des protéines arrivant et le quittant. Malgré le fait que ses protéines diffusent facilement dans le nucléoplasme, la taille relative des domaines SC35 reste constante (114). Ces taux d’échange pourraient être régulés à travers des cycles de phosphorylation/déphosphorylation. Par exemple, la phosphorylation des facteurs d’épissages SR a été montrée nécessaire pour adresser les protéines SR aux domaines SC35 (99).
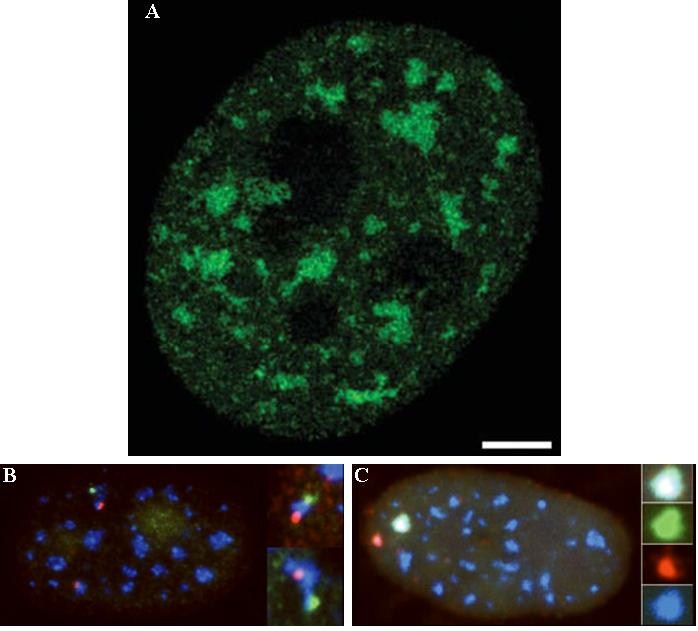
Figure : Relation entre les domaines SC35 et les gènes cellulaires. A. Visualisation
d’YFP-SC35 par microscopie confocale à haute résolution (système OMX) (140). Les
domaines SC35 ont un aspect de taches dans le noyau. Echelle : 2μm. B. Un même domaine
SC35 (en bleu) est associé avec les gènes de collagène1 A1 et A2 (vert et rouge
respectivement), qui sont sur des chromosomes différents. C. Les ARN de collagène1 A1 et
A2 sont localisés au sein d’un domaine SC35 (d’après (63)).
Les protéines SR
Les protéines SR sont des protéines possédant au moins un domaine riche en arginine et sérine (domaine RS), et sont parmi les constituants les plus abondants des domaines SC35. Elles peuvent être classées en trois catégories, les protéines SR classiques, les protéines SR additionnelles et les protéines SR apparentées. Les protéines SR classiques, auxquelles appartiennent les protéines SC35 et ASF/SF2, contiennent au moins un domaine RRM (motif de reconnaissance à l’ARN) en C terminal suivi d’un domaine RS riche en arginine et sérine. Le domaine RS favorise les interactions protéine-protéine afin de faciliter l’assemblage du splicéosome. Le domaine RS peut aussi entrer en contact directement avec le préARNm via le point de branchement (129). De plus, ce domaine agit comme un signal de localisation nucléaire (NLS), et permet l’adressage de ces protéines aux domaines SC35. Les protéines SR additionnelles sont des protéines contenant les mêmes domaines que les protéines SR classiques, mais dans un ordre différent. Ces deux catégories de protéines SR sont impliquées dans les mécanismes d’épissage de plusieurs gènes cellulaires. Enfin, la dernière catégorie de protéines SR apparentées, dont fait partie Son, est constituée par les protéines contenant un domaine RS et un autre domaine requis pour leur activité. On trouve dans cette dernière catégorie des protéines impliquées dans l’épissage mais ayant aussi d’autres fonctions.
L’épissage chez les eucaryotes résulte de l’activité du splicéosome, qui est un complexe composé de cinq snRNP et plusieurs facteurs protéiques qui reconnaissent les sites consensus d’épissage localisé en 5’ et en 3’ des séquences introniques. Cependant, la présence de sites d’épissage n’est généralement pas suffisante pour l’assemblage d’un splicéosome fonctionnel, et des séquences régulatrices connues sous le nom d’ESE/ISE (Exon Splicing Enhancer/ Intron Splicing Enhancer) et ESS/ISS (Exon Splicing Silencer/ Intron Splicing Silencer) sont nécessaires.
Ces éléments régulateurs sont impliqués dans l’épissage constitutif et alternatif en fixant des facteurs d’épissage. En particulier, la liaison des protéines SR aux ESE agit comme une barrière afin de prévenir l’élimination d’exons (70). Deux modèles ont été proposés pour expliquer ce phénomène. Les protéines SR peuvent faciliter le recrutement du tri-snRNP U4/U6.U5 au pré-splicesosome (120), ou elles peuvent interférer avec l’activité de hnRNP A/B au niveau de la sélection du site d’épissage. En effet, un excès de hnRNP A/B favorise la sélection de sites d’épissage 5’ distants, tandis qu’un excès de protéines SR permet l’utilisation de sites d’épissage 5’ proximaux (47). Il est intéressant de noter que les protéines SR individuelles peuvent parfois avoir un effet sur la sélection des sites d’épissage, avec par exemple ASF/SF2 et SC35 jouant un rôle dans la régulation de l’épissage alternatif des préARNm de la β-tropomyosine et de l’hormone de croissance humaine.
Les protéines SR ont aussi un rôle dans la transcription des gènes. Il a récemment été décrit que SC35 promeut l’élongation par l’ARN polymérase II d’un sous-ensemble de gènes, confirmant l’existence d’un couplage entre la transcription et l’épissage, et plus surprenant, montrant que ce couplage peut-être bidirectionnel (100). Cet effet s’expliquerait par le fait que SC35 interagit non seulement avec le domaine C-terminal de l’ARN polymérase II, comme d’autre protéines SR, mais aussi avec CDK9 (cyclin dependent kinase 9), qui phosphoryle le facteur d’élongation transcriptionnel P-TEFb (86).
Enfin, plusieurs études montrent que l’activité et la localisation des protéines SR sont régulées par la phosphorylation de résidus sérine. Par exemple, la phosphorylation du domaine RS de ASF/SF2 augmente ses interactions avec les autres protéines SR, alors que la dé-phosphorylation de RS est requise pour le processus d’épissage (22). De même, un état hypo-phosphorylé de la protéine SR est nécessaire pour l’interaction avec le récepteur d’export nucléaire des ARNm TAP/NXF1, tandis qu’une re-phosphorylation du domaine RS de la protéine dans le cytoplasme est nécessaire pour que la protéine puisse retourner dans le noyau (36).
En conclusion, les protéines SR jouent un rôle dans plusieurs étapes du métabolisme des ARNm : la transcription, l’épissage, et enfin l’export de certains ARNm vers le cytoplasme.
1.4 Les corps de Cajal
Les corps de Cajal ont été identifiés par Ramon y Cajal, qui a obtenu en 1906 le prix Nobel avec Camillo Golgi pour ses recherches sur l’architecture cellulaire du système nerveux. En utilisant une technique d’imprégnation à l’argent, Cajal a trouvé à l’intérieur du noyau des cellules nerveuses un petit corps rond d’un diamètre de 0,3 à 1 µm, qu’il a appelé corps accessoire (19). Le même genre de corps a été ensuite identifié dans les cellules de mammifères, d’amphibien, d’insectes et dans les plantes. C’est en découvrant un marqueur commun à tout ces corps, la coïline, qu’il a été décidé d’uniformiser le nom en corps de Cajal (118).Après la découverte de la coïline dans les cellules humaines, cette protéine est devenue le marqueur de référence pour les corps de Cajal dans toutes les cellules de mammifères. Malgré le fait que l’on se serve de cette protéine comme marqueur, peu de chose sont connues sur sa fonction biochimique dans les corps de Cajal. La coïline se lie au moteur de survie des neurones (SMN) et à plusieurs protéines Sm (constituant le snRNP) suggérant qu’elle aurait un rôle dans l’assemblage et la modification des snRNP constituant le spliceosome (156). De plus, la suppression de la coïline chez la souris détruit complètement les corps de Cajal, amenant un phénotype partiellement létal, et des souris survivantes présentant des défauts importants en termes de fertilité et de fécondité (147).
En dehors de la coïline, les corps de Cajal sont composés en majorité de snRNP, en particulier les snRNP U1, U2, U4, U5 et U6 (139). Parce que l’épissage en lui-même n’intervient pas dans les corps de Cajal, il est probable que ces deniers jouent un rôle dans la formation et la modification des snRNP avant leur recrutement aux sites de transcription. La biogénèse des snRNP est un processus complexe comportant des étapes cytoplasmiques et nucléaires. En bref, la transcription des snRNA qui a lieu dans le noyau, est suivie par leur exportation dans le cytoplasme. L’extrémité 5’ est alors tri-méthylée, et chaque snRNA est assemblé en complexe avec sept protéines Sm conservées, nécessaires pour les réactions d’épissage. Finalement, ces complexes retournent dans le noyau, et se concentrent dans les corps de Cajal, avant d’être dirigés dans les domaines SC35, puis éventuellement au niveau de gènes actifs où ils vont jouer un rôle essentiel dans l’épissage (134). Les corps de Cajal jouent probablement un rôle dans la modification et la maturation finale des snRNP, par exemple en assemblant le tri-snRNP U4/U6-U5 (141). Il a aussi été proposé que le recyclage des snRNP puisse prendre place dans les corps de Cajal.
De plus, les protéines TR et TERT (composants de la télomérase) sont localisées dans les corps de Cajal dans des lignées de cellules cancéreuses, suggérant que les corps de Cajal pourraient avoir un rôle dans l’assemblage et la maturation de la télomérase (159).
J’ai précédemment présenté 3 modèles d’assemblage des corps nucléaires. Il a été montré que le modèle stochastique est le mode d’assemblage des corps de Cajal (75). Dans cette étude, les différents composants des corps de Cajal ont été fixés artificiellement à un locus spécifique dans des cellules Hela. L’immobilisation de n’importe lequel de ces composants était suffisante pour initier le recrutement de la plupart (si ce n’est tous) des autres composants des corps de Cajal. Le nouveau corps a une taille, une forme et une composition similaire au corps endogène et ses composants présentent une cinétique semblable. Il reste cependant à déterminer si ce phénomène n’était pas causé par le recrutement d’un corps de Cajal déjà formé. Par ailleurs, il n’a pas été démontré que les corps ainsi formé sont fonctionnels.
Concernant la mobilité des composants des corps de Cajal, diverses expériences de photo-blanchiment montrent, comme pour les autres corps nucléaires, un taux d’échange rapide des composants entre le corps nucléaire et le nucléoplasme. Cependant, les mouvements sont un peu plus lents que pour les autres corps nucléaires, le temps de demi-recouvrement étant de quelques minutes (40).
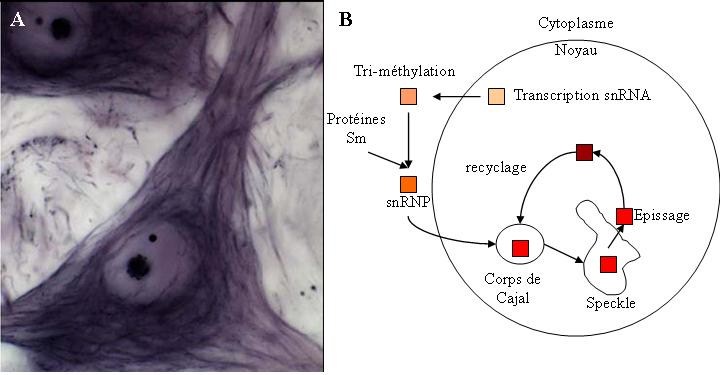
Figure : Visualisation et fonction des corps de Cajal. A. Coloration d’un neurone en utilisant la méthode de Cajal (réduction au nitrate d’argent). Le nucléole, le corps de Cajal et les neurofibrilles sont visibles ; B. Schéma explicatif de la fonction du corps de Cajal : le snRNA, synthétisé dans le noyau, est exporté vers le cytoplasme, où il est transformé en snRNP par des modifications post-traductionnelles et l’ajout de protéines Sm. Les snRNP repassent ensuite dans le noyau pour atteindre les corps de Cajal, où ils sont maturés, avant d’être recrutés et utilisés par le spliceosome en passant par les domaines SC35. Une fois utilisé, les snRNP peuvent être recyclés en repassant par les corps de Cajal, et ainsi être de nouveau utilisés (d’après (80)).
1.5 Les paraspeckles
Les paraspeckles sont de nouveaux corps nucléaires ayant été identifiés en 2002 (55). Une analyse protéomique du nucléole humain a permis d’identifier 271 nouvelles protéines, et environ 30% d’entre elles étaient nouvellement décrites. Or, une analyse supplémentaire à permis de montrer que quelques protéines n’étaient pas enrichies dans le nucléole, mais se localisent dans des foyers nucléoplasmiques. Ces nouveaux corps ont été nommés paraspeckles car ils sont observés dans l’espace inter-chromatinien à proximité des domaines SC35. La protéine servant de marqueur de référence de ces corps a été nommée « paraspeckles protein 1 » (PSPC1). Dans des cellules privées d’activité de transcription, PSPC1 est relocalisée dans le compartiment périnucléolaire, et des expériences de photo-blanchiment ont montré que PSPC1 présente un trafic continu entre le nucléole et les paraspeckles, expliquant ainsi sa présence dans le nucléole (55).Les paraspeckles sont des corps riches en ARN, de taille comprise entre 0.5 et 1 µm de diamètre, de taille irrégulière et inégalement répartis dans le noyau (23). Quelques protéines se trouvent également enrichies dans les paraspeckles (dont p54nrb et SFPQ), et la plupart d’entre elles ont un rôle dans la transcription et/ou la maintenance des ARN. Il est intéressant de noter qu’un des composants fondamentaux des paraspeckles est l’ARN non codant (ARNnc) NEAT1, qui est nécessaire à leur formation et leur maintien (124).
Des études récentes ont montré que la synthèse de transcrits de NEAT1 est suffisante pour initier la formation des paraspeckles. Une fois transcrits, les ARNnc NEAT1 forment des complexes avec les protéines des paraspeckles, généralement avant que l’ARN ne puisse diffuser à travers le nucléoplasme. Le paraspeckle est donc un ensemble dynamique composé d’ARN et de protéines formant une construction solide. Comme dans les autres corps nucléaires, il présente un haut taux d’échange entre la fraction de protéines présente dans le corps et le nucléoplasme (89). Ainsi, les trois protéines principales des paraspeckles montrent une fraction immobile de 30 à 40% et le taux de demi-recouvrement après photo-blanchiment de ces protéines est d’environ 5 à 10 secondes. Il a de plus été montré que les paraspeckles ne sont formés que lorsqu’il y a transcription active de NEAT1 (89). Malgré leur confinement à proximité du locus NEAT1, les paraspeckles bougent et présentent des événements de fusion et de fission, jusqu’à former des grappes autour de ce locus.
Le rôle principal des paraspeckles est de retenir des structure d’ARN double brin hyper-édités formé par des répétions inversées. L’édition de A en I est une conversion des Adénosines en Inosines réalisée par une déaminase (ADAR1) qui reconnaît les longs ARN double brin. Cette séquence d’ARN hyper-édités est alors reconnue par p54nrb, qui va la retenir dans les paraspeckles en interagissant avec PSPC1 et SFPQ. La rétention d’un ARNm pourrait survenir suite à un stress ou en réponse à une infection virale. Il est aussi possible que les paraspeckles jouent un rôle dans la différenciation cellulaire, probablement en altérant l’expression de gènes clés par la rétention des ARN.
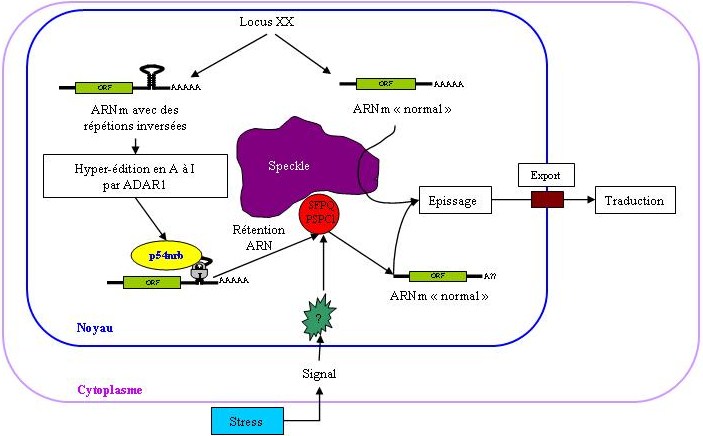
Figure : Fonction des paraspeckles. La transcription d'un gène (ici le locus XX) peut donner deux types d'ARN. Un ARN "normal", qui va être épissé, exporté puis traduit. Ou un ARN des répétitions inversées en 3'. Ces répétitions vont être reconnues puis hyper-édité en A à I (Adénosine en Inosine) par ADAR1. Une fois hyper-éditées, ces séquences vont être reconnus et retenus par p54nrb. Cette protéine va alors intéragir avec PSF/SFPQ et/ou PSPC1 dans les paraspeckles, et causé la rétention de cet ARN dans les paraspeckles. Seul une coupure de l'ARN au niveau des sites hyper-édités peut alors libérer l'ARN, coupure pouvant être causé par un stress cellulaire, par exemple. L'ARN libéré va alors suivre la voie d'un ARN "normal" (d'après (57)).
1.6 Les centres de transcription
En 1993, il a été proposé que la transcription des gènes ait lieu dans des centres spécialisés caractérisés par une forte concentration en certaines formes d’ARN polymérase et qui ont été appelés centres de transcription (Transcription Factories) (68).Chaque centre de transcription est défini comme un site nucléaire contenant au moins deux complexe d’ARN polymérase actifs (29). Dans des cellules Hela, un centre de transcription typique contient environ huit enzymes engagées sur huit brins d’ADN. La fonction de ces centres serait de faciliter la transcription d’ARN en concentrant au maximum les réactifs et les substrats au même endroit. Ainsi, dans un noyau de cellules Hela la concentration en ARN polymérase est mille fois plus importante dans les centres de transcription, où se trouvent la majorité des ARN naissants, par apport au nucléoplasme. Le cœur des centres de transcription est polymorphique, avec un diamètre d’environ 87 nm (49). L’existence de centres de transcription dans lesquels les gènes actifs co-localisent implique que ce sont les gènes qui se déplacent vers et à l’intérieur du centre, contrairement à la vision classique qui suppose que ce soient les complexes enzymatiques qui « scannent » les fibres de la chromatine, comme une locomotive sur des rails. Ce modèle implique aussi que l’ARN polymérase engagée dans la transcription soit immobilisée dans le centre (30). Bien sûr, les mouvements sont relatifs et la polymérase peut être fixée au foyer de transcription, mais le foyer en lui-même peut bouger à l’intérieur du noyau.
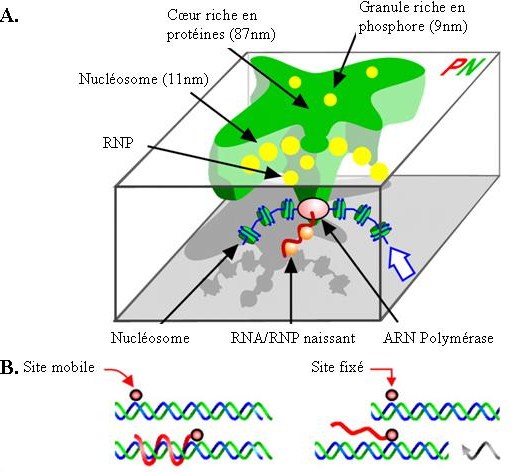
Figure : Organisation d'un centre de transcription. A. Le centre de transcription est
composé d’un coeur de protéines (vert), avec des RNP et nucléosome (jaune) à sa surface.
L’ARN polymérase est fixée sur le coeur du centre de transcription, et transcrit l’ADN
(d’après (50)). B. Le fait que la polymérase soit fixée évite que l’ARN s’enroule autour de la
double hélice d’ADN (d’après (29)).
2 Relation entre virus et corps nucléaires
Les corps nucléaires ont des rôles spécifiques dans le noyau de la cellule, et il est légitime de penser que les virus détournent la fonction première de ces corps afin de les neutraliser ou de s’en servir pour leur propre transcription ou leur réplication.2.1 Virus et nucléole
Le nucléole est un corps nucléaire contrôlant de nombreux mécanismes utiles pour les virus. Ainsi, détourner tout ou une partie des fonctions du nucléole permet à ceux-ci, soit de prendre le contrôle de certains mécanismes, soit de favoriser leur réplication (revue en (59)).Le virus adéno-associé (AAV) est un virus ayant besoin d’un virus auxiliaire pour sa réplication, comme l’adénovirus. Lorsqu’il y a co-infection de ces deux virus, les protéines Rep et Cap de l’AAV sont localisées dans le nucléole (13, 115). De plus, la protéine nucléolaire B23 et la nucléoline joueraient un rôle dans l’assemblage de la capside de l’AAV dans le nucléole, et la protéine virale Rep jouerait un rôle dans l’export de la capside complète du nucléole vers le noyau, au niveau des sites d’encapsidation (13, 115, 152).
Lors de l’infection par HSV-1, et tant que la réplication est active, les protéines nucléoline et UBF (Upstream Binding Factor) sont redistribuées aux compartiments de réplication virale, où elles sont co-localisées avec la protéine virale de liaison à l’ADN ICP8 (20). Il a de plus été mis en évidence que l’expression et la réplication du virus diminuent dans des cellules sans nucléoline (20). UBF participe à la réplication virale, tandis que la nucléoline est nécessaire au virus pour l’expression de ses gènes ainsi qu’à sa réplication (20). De plus, il a été mis en évidence que la protéine virale UL27 induit la relocalisation de la nucléoline dans le cytoplasme, même lorsqu’elle est exprimée seule, soulignant la relation entre HSV et le nucléole (12). Un autre virus interagissant avec le nucléole est le papillomavirus (HPV). La nucléoline faciliterai la transcription de l’oncogène viral E6/E7, et contrôlerait ainsi l’effet tumoral induit par HPV-18 (61). La protéine adénovirale pX, requise pour l’assemblage des centres de réplication viraux transite par le nucléole, et induit la relocalisation de trois protéines nucléolaire, B23, l’ARN polymérase I et UBF qui vont se localiser dans les centres de réplication de l’adénovirus pour prendre part à la réplication de l’ADN viral (83). L’adénovirus code aussi une protéine « core V » qui est ciblée au nucléole, et qui induit la redistribution de la nucléoline et B23 vers le cytoplasme lorsqu’elle est exprimée seule (92).
Dans le cas des virus à ARN, le virus de l’hépatite C (HCV) exprime une protéine non-structurale 5B (NS5B), l’ARN polymérase virale, qui relocalise la nucléoline dans le cytoplasme (66), où elle va se fixer aux IRES (Internal Ribosome Entry Site) de l’ARN viral afin de permettre la traduction de ces ARNm (88). L’interaction entre la nucléoline et NS5B est nécessaire à l’oligomérisation de cette dernière, oligomérisation qui est un pré-requis pour son activité et donc nécessaire pour une réplication efficace du génome d’HCV (66). Le virus de l’Hépatite Delta (HDV) qui est un virus satellite du virus de l’Hépatite B (HBV), est aussi associé au nucléole. L’antigène spécifique de ce virus (HDAg) se localise dans le nucléole grâce à son interaction avec la nucléoline. Par ailleurs, le nucléole est aussi le site où a lieu la réplication du génome viral (84).
Le nucléole est également impliqué dans l’exportation des ARNm viraux d’HTLV-1 (virus T-lymphotropique humain). La protéine virale Rex, associée avec CRM1 dans le nucléole, active l’export des ARNm viraux. La protéine virale p30 se fixe à Rex, et une sur-expression de p30 va séquestrer Rex-CRM1, diminuant ainsi l’export des ARNm viraux (62).
HIV-1 (Human Immunodeficiency Virus) est un lentivirus ayant des interactions à plusieurs niveaux avec le nucléole et ses composants. Ainsi, la nucléoline, dont une partie est membranaire, est probablement impliquée dans l’attachement du virus à la cellule via gp120 (122). Plusieurs protéines et ARNm viraux transitent par le nucléole, dont Tat et Rev, qui interagissent avec B23. Lors d’une infection à HIV, la protéine virale Tat est essentielle pour la régulation de la transcription virale. Tat est localisée dans le nucléole avec B23, et recrute le facteur de transcription positif d’élongation P-TEFb, qui permet d’activer la transcription des ARN d’HIV (15). Enfin, la nucléoline pourrait être impliquée dans l’incorporation de gag dans les nouveaux virions.
En conclusion, les virus détournent les différentes protéines et fonctions nucléolaire pour leur propre usage, mais de façon différente, afin de faciliter l’export ou la transcription des ARNm viraux par exemple.
2.2 Virus et corps PML
Les corps PML sont les structures pour lesquelles les interactions avec les virus ont été le plus étudiées. Ces études ont montré que les corps PML ont globalement un rôle anti-viral, notamment en induisant la répression des gènes viraux (revue en (52)).Les virus herpès font partis des virus les plus connus interagissant avec les corps PML, et ayant la capacité de les détruire. En particulier, de nombreuses études ont montré que la protéine très précoce ICP0 de l’HSV-1 est suffisante pour détruire les corps PML (53). Cette protéine, qui est une des premières à être exprimée, se localise très tôt au niveau des corps PML au sein desquels elle induit la dégradation des formes sumoylées de PML et SP100 grâce à son activité E3 ubiquitine ligase (50). L’hypothèse actuelle est que les protéines des corps PML exercent une activité antivirale, notamment en s’accumulant à proximité des génomes viraux entrant dans le noyau. La destruction de PML par ICP0 contribuerait alors à dé-réprimer les génomes viraux et à induire le cycle réplicatif (51). D’autres virus herpès codent des protéines ayant des propriétés similaires à ICP0. Notamment, le cytomégalovirus humain (HCMV) code pour une protéine constituant les particules virales, pp71, qui est colocalisée aux temps précoces de l’infection avec les corps PML, où elle induit la dégradation de DAXX. Cette protéine stimule aussi l’expression de gènes viraux, dont la protéine IE72 qui est localisée au niveau des corps PML, qu’elle va perturber en dé-sumoylant la protéine PML, puis en détruisant les corps PML (1). La protéine EBNA1 du virus d’Epstein-Barr (EBV) peut aussi perturber les corps PML. EBNA1 augmente l’association d’UPS7 (ubiquitine protéase) et CK2 (kinase) avec PML, ce qui va conduire à une dé-ubiquitinylation et une phosphorylation de cette dernière, puis à sa dégradation (133). De plus, comme dans le cas d’HSV-1, le génome d’EBV co-localise avec les corps PML à des temps précoces, mettant ainsi en évidence la relation étroite entre ce virus et ce corps nucléaire (4).
D’autre virus à ADN perturbent aussi les corps PML. Les protéines de l’Adénovirus E1A et E4orf3 sont associées avec les corps PML, et E4orf3 est suffisante pour les perturber en induisant leur relocalisation en structure « track-like » (24). Dans les temps plus tardifs de l’infection, la protéine pIX va relocaliser de façon stable la protéine PML dans des inclusions nucléaires, et ainsi contribuer à augmenter la prolifération virale (119). La protéine de la capside du papillomavirus L2 est trouvé localisée dans les corps PML, où elle délivre le génome viral (32). Ainsi, contrairement aux autres virus à ADN qui détruisent les corps PML, le présence du génome viral au sein des corps PML permet la transcription virale et l’assemblage des foyers de réplication (144).
Les virus à ARN (et rétrovirus) interagissent aussi avec les corps PML. L’oncoprotéine Tax du virus HTLV-1 altère la localisation de la protéine Int-6, qui est normalement associée au corps PML (35). Int6 est un composant du complexe d’initiation de la traduction (eIF-3), et cette liaison va l’amener dans le cytoplasme, mais ne va pas perturber le corps nucléaire en lui même. Ce phénomène pourrait permettre à d’autre composants des corps PML d’activer l’expression de gènes sous le contrôle de Tax (6). Les infections par le virus de la choriomeningite lymphocytaire (LCMV), et plus précisément sa protéine Z, causent une redistribution de la protéine PML des corps nucléaires au cytoplasme ainsi qu’une diminution de l’expression d’eIF-4E et de la cycline D1. Pris ensemble, ces résultats suggèrent que ces interactions permettent au virus de ralentir la croissance cellulaire, ce qui constituerait une stratégie pour établir une infection chronique (21).
En conclusion, la plupart des virus perturbent les corps PML. Cet événement a lieu car les corps PML présentent une activité antivirale, mais contiennent aussi de nombreux facteurs qui peuvent être utilisés par les virus pour favoriser leur transcription et leur réplication.
2.3 Virus et domaines SC35
De nombreux virus utilisent les domaines SC35 afin de favoriser leur transcription et/ou épissage. Parmi ces virus, les virus de l’Herpès ont une relation spéciale avec les domaines SC35. En effet, HSV-1 assemble ses centres de réplication précoces à la surface des domaines SC35. Ceci permet une réorganisation favorisant l’export des ARN viraux tardifs (26). Il a de plus été mis en évidence que la protéine virale ICP27 interagit avec le complexe d’export des ARNm TAP/NXF1, et que les protéines SR SRp20 et 9G8 participent à l’export des ARNm viraux (48). Dans le cas du virus de la varicelle (VZV), il y a une interaction entre la protéine virale précoce IE4 avec les protéines SR ASF/SF2, 9G8 et SRp20. Il y a aussi une forte interaction avec la kinase SRPK1 et le complexe d’export des ARNm TAP/NXF1. IE4 interagit probablement avec les protéines SR (ayant elles même reconnu des ARN viraux), puis avec le complexe d’export TAP/NXF1 afin de passer dans le cytoplasme. Une fois le complexe dans le cytoplasme, SRPK1 dé-phosphoryle les protéines SR, qui libèrent alors l’ARN viral, puis repassent dans le noyau avec IE4. Un défaut des protéine SR ou de TAP/NXF1 résulte en la diminution de l’expression des protéines virales (111). La protéine SM du virus Epstein Barr (EBV) stabilise, participe à l’épissage et facilite l’export des ARNm viraux. La protéine SRp20 interagit avec SM, et cette interaction est importante pour l’épissage alternatif. Ainsi, un défaut de SRp20 inhibe l’épissage par la protéine SM (145).D’autre virus à ADN interagissent aussi avec les domaines SC35, dont le papillomavirus et l’adénovirus. Dans le cas du papillomavirus, l’infection des cellules entraine une surexpression de protéines SR (dont ASF/SF2, SC35 et SRp20), qui sont nécessaires pour l’épissage ou l’export des ARN viraux (94). Dans le cas de l’adénovirus, les protéines SR ASF/SF2, SC35 et 9G8 permettent l’épissage ou l’export des transcrits viraux tardifs qui s’accumulent dans le noyau dans les phases tardives de l’infection (65).
Le virus de la grippe (Influenza B) possède un génome à ARN qui se réplique et transcrit ses ARN dans le noyau de la cellule hôte. Il a été montré que la protéine NS1 est colocalisée avec SC35 au temps précoce de l’infection, avant d’être relocalisée au cytoplasme. Cette accumulation de NS1 dans les domaines SC35 suggère un rôle important de cette protéine virale dans la biogénèse de certains ARN viraux ayant un impact sur la réplication du virus (125). Il reste cependant à approfondir quelles sont les relations entre le virus et ce corps nucléaires, et plus précisément quelles sont les ARN ou protéines virales transitant par les domaines SC35.
Plusieurs rétrovirus montrent des interactions avec les domaines SC35 ou leur composant. Le premier et plus connu est HIV. La sur- ou sous-expression de hnRNP ou de protéines SR favorise l’utilisation de certains sites d’épissage au détriment d’autres et diminue le taux de transcription des ARN viraux. Ainsi, la surexpression de hnRNPA1 diminue la production de virions d’un facteur 100 sans affecter la viabilité de la cellule (71). Le virus du sarcome de Rous (RSV) nécessite pour sa réplication un certain ratio entre ses ARN épissés et non-épissés. Une séquence spécifique dans les ARN viraux, appelée le régulateur négatif d’épissage permet de garder un ratio optimal. Or, il a été observé qu’un certain nombre des protéines SR se fixent sur ce régulateur, plus particulièrement ASF/SF2 et SC35, et pourraient ainsi intervenir dans la régulation de ce ratio (95). HTLV-1 et 2 interagissent aussi avec les domaines SC35. Plus précisément, les protéines Tax de ces virus sont localisées dans des structures, qui se superposent en partie au domaine SC35 (127). L’association de Tax avec les domaines SC35 survient lorsque Tax est sumoylée alors que son ubiquitinylation conduit à une localisation cytoplasmique, permettant l’activation de la voie NFκB (107).
En conclusion, les domaines SC35 permettent aux virus de contrôler finement leurs réactions d’épissage ainsi que l’export de leur ARNm, optimisant ainsi leur réplication ou le contrôle de leur génome dans le cas des rétrovirus.
| Virus | Description |
| HSV | Fusion des CR viraux à la surface des domaines SC35 : exportation ARN tardif. SRp20 et 9G8 participent à l’exportation des ARN viraux. |
| VZV | ASF/SF2, 9G8 et SRp20 reconnues par IE4 et les ARNm viraux : exportation. SRPK1 libère le complexe, et IE4 peut alors repasser dans le noyau. |
| EBV | SRp20 interagit avec la protéine virale SM dans l’épissage des ARNm viraux. |
| HPV | ASF/SF2, SC35 et SRp20 : impliquées dans l’épissage ou l’exportation des ARN viraux. |
| Adénovirus | ASF/SF2, SC35 et 9G8 : impliquées dans l’épissage ou l’exportation des ARN viraux tardifs. |
| Influenza-B | NS1 et SC35 sont co-localisées aux temps précoces de l’infection. |
| HIV-1 | Le taux des protéines SR et hnRNP régule l’épissage des ARN viraux. |
| RSV | ASF/SF2 et SC35 se fixent sur le régulateur négatif d’épissage du virus. |
| HTLV-1 et 2 | Association de Tax aux domaines SC35 : activation de gènes cellulaires et viraux. |
2.4 Virus et corps de Cajal
Pour le moment, seul l’adénovirus a été décrit comme interagissant avec les corps de Cajal (72). Dans cette étude, il est montré que lors d’une infection, ces corps sont distribués en rosettes, localisées autour des centres de réplication viraux. Ces rosettes auraient un rôle dans la réplication du virus, et plus particulièrement dans l’expression des gènes tardifs. En effet, un défaut d’expression de la coïline résulte en la diminution d’environ 30% de l’expression des gènes viraux tardifs. L’hypothèse émise est que la réorganisation en rosettes pourrait servir à optimiser la transcription et l’épissage, probablement en augmentant l’aire d’échange entre le centre de réplication viral et les sites de transcription et d’épissage.2.5 Virus et paraspeckles
Les paraspeckles ont pour fonction connue la rétention d’ARN hyper-édités, et leurs composants transitent par le nucléole. Etonnamment, une analyse du protéome nucléolaire des cellules infectées par le virus de l’Influenza A montre que les principaux composants des paraspeckles sont perturbés par cette infection (46). Il est ainsi observé une diminution d’un facteur 2 pour les trois protéines constitutives des paraspeckles (PSPC1, p54nrb et SFPQ) dans le nucléole et une augmentation d’un facteur 3 d’ADAR1, qui peut être responsable d’hyper-mutation dans l’ARN du virus. Le virus pourrait ainsi se servir de certaines fonctions des paraspeckles (mutation) tout en évitant la rétention de ses ARN.Bibliographie
1. Ahn, J. H., and G. S. Hayward. 2000. Disruption of PML-associated nuclear bodies by IE1 correlates with efficient early stages of viral gene expression and DNA replication in human cytomegalovirus infection. Virology 274:39-55.
2. Alazard-Dany, N., A. Nicolas, A. Ploquin, R. Strasser, A. Greco, A. L. Epstein, C. Fraefel, and A. Salvetti. 2009. Definition of herpes simplex virus type 1 helper activities for adeno-associated virus early replication events. PLoS Pathog 5:e1000340.
3. Albiez, H., M. Cremer, C. Tiberi, L. Vecchio, L. Schermelleh, S. Dittrich, K. Kupper, B. Joffe, T. Thormeyer, J. von Hase, S. Yang, K. Rohr, H. Leonhardt, I. Solovei, C. Cremer, S. Fakan, and T. Cremer. 2006. Chromatin domains and the interchromatin compartment form structurally defined and functionally interacting nuclear networks. Chromosome Res 14:707-33.
4. Amon, W., R. E. White, and P. J. Farrell. 2006. Epstein-Barr virus origin of lytic replication mediates association of replicating episomes with promyelocytic leukaemia protein nuclear bodies and replication compartments. J Gen Virol 87:1133-7.
5. Andersen, J. S., Y. W. Lam, A. K. Leung, S. E. Ong, C. E. Lyon, A. I. Lamond, and M. Mann. 2005. Nucleolar proteome dynamics. Nature 433:77-83.
6. Ariumi, Y., T. Ego, A. Kaida, M. Matsumoto, P. P. Pandolfi, and K. Shimotohno. 2003. Distinct nuclear body components, PML and SMRT, regulate the trans-acting function of HTLV-1 Tax oncoprotein. Oncogene 22:1611-9.
7. Atchison, R. W., B. C. Casto, and W. M. Hammon. 1965. Adenovirus-Associated Defective Virus Particles. Science 149:754-6.
8. Audas, T. E., M. D. Jacob, and S. Lee. 2012. Immobilization of proteins in the nucleolus by ribosomal intergenic spacer noncoding RNA. Mol Cell 45:147-57.
9. Berger, G., S. Durand, C. Goujon, X. N. Nguyen, S. Cordeil, J. L. Darlix, and A. Cimarelli. 2011. A simple, versatile and efficient method to genetically modify human monocyte-derived dendritic cells with HIV-1-derived lentiviral vectors. Nat Protoc 6:806-16.
10. Bernard, D., K. V. Prasanth, V. Tripathi, S. Colasse, T. Nakamura, Z. Xuan, M. Q. Zhang, F. Sedel, L. Jourdren, F. Coulpier, A. Triller, D. L. Spector, and A. Bessis. 2010. A long nuclear-retained non-coding RNA regulates synaptogenesis by modulating gene expression. Embo J 29:3082-93.
11. Berns, K. I., and C. Giraud. 1996. Biology of adeno-associated virus. Curr Top Microbiol Immunol 218:1-23.
12. Bertrand, L., and A. Pearson. 2008. The conserved N-terminal domain of herpes simplex virus 1 UL24 protein is sufficient to induce the spatial redistribution of nucleolin. J Gen Virol 89:1142-51.
13. Bevington, J. M., P. G. Needham, K. C. Verrill, R. F. Collaco, V. Basrur, and J. P. Trempe. 2007. Adeno-associated virus interactions with B23/Nucleophosmin: identification of sub-nucleolar virion regions. Virology 357:102-13.
14. Bolzer, A., G. Kreth, I. Solovei, D. Koehler, K. Saracoglu, C. Fauth, S. Muller, R. Eils, C. Cremer, M. R. Speicher, and T. Cremer. 2005. Three-dimensional maps of all chromosomes in human male fibroblast nuclei and prometaphase rosettes. PLoS Biol 3:e157.
15. Brady, J., and F. Kashanchi. 2005. Tat gets the "green" light on transcription initiation. Retrovirology 2:69.
16. Brister, J. R., and N. Muzyczka. 2000. Mechanism of Rep-mediated adeno-associated virus origin nicking. J Virol 74:7762-71.
17. Brown, J. M., J. Green, R. P. das Neves, H. A. Wallace, A. J. Smith, J. Hughes, N. Gray, S. Taylor, W. G. Wood, D. R. Higgs, F. J. Iborra, and V. J. Buckle. 2008. Association between active genes occurs at nuclear speckles and is modulated by chromatin environment. J Cell Biol 182:1083-97.
18. Buller, R. M., J. E. Janik, E. D. Sebring, and J. A. Rose. 1981. Herpes simplex virus types 1 and 2 completely help adenovirus-associated virus replication. J Virol 40:241-7.
19. Cajal, R. 1910. El nucleo de las celulas piramidales del cerebro humano y de algunos mamiferos. Trab Lab Invest Biol Univ Madrid 8:p. 27-62.
20. Calle, A., I. Ugrinova, A. L. Epstein, P. Bouvet, J. J. Diaz, and A. Greco. 2008. Nucleolin is required for an efficient herpes simplex virus type 1 infection. J Virol 82:4762-73.
21. Campbell Dwyer, E. J., H. Lai, R. C. MacDonald, M. S. Salvato, and K. L. Borden. 2000. The lymphocytic choriomeningitis virus RING protein Z associates with eukaryotic initiation factor 4E and selectively represses translation in a RING-dependent manner. J Virol 74:3293-300.
22. Cao, W., S. F. Jamison, and M. A. Garcia-Blanco. 1997. Both phosphorylation and dephosphorylation of ASF/SF2 are required for pre-mRNA splicing in vitro. Rna 3:1456-67.
23. Cardinale, S., B. Cisterna, P. Bonetti, C. Aringhieri, M. Biggiogera, and S. M. Barabino. 2007. Subnuclear localization and dynamics of the Pre-mRNA 3' end processing factor mammalian cleavage factor I 68-kDa subunit. Mol Biol Cell 18:1282-92.
24. Carvalho, T., J. S. Seeler, K. Ohman, P. Jordan, U. Pettersson, G. Akusjarvi, M. Carmo-Fonseca, and A. Dejean. 1995. Targeting of adenovirus E1A and E4-ORF3 proteins to nuclear matrix-associated PML bodies. J Cell Biol 131:45-56.
25. Cervelli, T., J. A. Palacios, L. Zentilin, M. Mano, R. A. Schwartz, M. D. Weitzman, and M. Giacca. 2008. Processing of recombinant AAV genomes occurs in specific nuclear structures that overlap with foci of DNA-damage-response proteins. J Cell Sci 121:349-57.
26. Chang, L., W. J. Godinez, I. H. Kim, M. Tektonidis, P. de Lanerolle, R. Eils, K. Rohr, and D. M. Knipe. 2011. Herpesviral replication compartments move and coalesce at nuclear speckles to enhance export of viral late mRNA. Proc Natl Acad Sci U S A 108:E136-44.
27. Cho, G., Y. Lim, and J. A. Golden. 2009. SUMO interaction motifs in Sizn1 are required for promyelocytic leukemia protein nuclear body localization and for transcriptional activation. J Biol Chem 284:19592-600.
28. Cmarko, D., P. J. Verschure, T. E. Martin, M. E. Dahmus, S. Krause, X. D. Fu, R. van Driel, and S. Fakan. 1999. Ultrastructural analysis of transcription and splicing in the cell nucleus after bromo-UTP microinjection. Mol Biol Cell 10:211-23.
29. Cook, P. R. 2010. A model for all genomes: the role of transcription factories. J Mol Biol 395:1-10.
30. Cook, P. R. 1999. The organization of replication and transcription. Science 284:1790-5.
31. Cremer, T., and M. Cremer. 2010. Chromosome territories. Cold Spring Harb Perspect Biol 2:a003889.
32. Day, P. M., C. C. Baker, D. R. Lowy, and J. T. Schiller. 2004. Establishment of papillomavirus infection is enhanced by promyelocytic leukemia protein (PML) expression. Proc Natl Acad Sci U S A 101:14252-7.
33. de Stanchina, E., E. Querido, M. Narita, R. V. Davuluri, P. P. Pandolfi, G. Ferbeyre, and S. W. Lowe. 2004. PML is a direct p53 target that modulates p53 effector functions. Mol Cell 13:523-35.
34. de Thé, M. Riviere, and W. Bernhard. 1960. [Examination by electron microscope of the VX2 tumor of the domestic rabbit derived from the Shope papilloma]. Bull Assoc Fr Etud Cancer 47:570-84.
35. Desbois, C., R. Rousset, F. Bantignies, and P. Jalinot. 1996. Exclusion of Int-6 from PML nuclear bodies by binding to the HTLV-I Tax oncoprotein. Science 273:951-3.
36. Ding, J. H., X. Y. Zhong, J. C. Hagopian, M. M. Cruz, G. Ghosh, J. Feramisco, J. A. Adams, and X. D. Fu. 2006. Regulated cellular partitioning of SR protein-specific kinases in mammalian cells. Mol Biol Cell 17:876-85.
37. Douar, A. M., K. Poulard, D. Stockholm, and O. Danos. 2001. Intracellular trafficking of adeno-associated virus vectors: routing to the late endosomal compartment and proteasome degradation. J Virol 75:1824-33.
38. Duan, D., P. Sharma, J. Yang, Y. Yue, L. Dudus, Y. Zhang, K. J. Fisher, and J. F. Engelhardt. 1998. Circular intermediates of recombinant adeno-associated virus have defined structural characteristics responsible for long-term episomal persistence in muscle tissue. J Virol 72:8568-77.
39. Duan, D., Z. Yan, Y. Yue, and J. F. Engelhardt. 1999. Structural analysis of adeno-associated virus transduction circular intermediates. Virology 261:8-14.
40. Dundr, M., M. D. Hebert, T. S. Karpova, D. Stanek, H. Xu, K. B. Shpargel, U. T. Meier, K. M. Neugebauer, A. G. Matera, and T. Misteli. 2004. In vivo kinetics of Cajal body components. J Cell Biol 164:831-42.
41. Dundr, M., and T. Misteli. 2011. Biogenesis of nuclear bodies. Cold Spring Harb Perspect Biol 2:a000711.
42. Dundr, M., and T. Misteli. 2001. Functional architecture in the cell nucleus. Biochem J 356:297-310.
43. Dundr, M., J. K. Ospina, M. H. Sung, S. John, M. Upender, T. Ried, G. L. Hager, and A. G. Matera. 2007. Actin-dependent intranuclear repositioning of an active gene locus in vivo. J Cell Biol 179:1095-103.
44. Dutheil, N., M. Yoon-Robarts, P. Ward, E. Henckaerts, L. Skrabanek, K. I. Berns, F. Campagne, and R. M. Linden. 2004. Characterization of the mouse adeno-associated virus AAVS1 ortholog. J Virol 78:8917-21.
45. Dye, B. T., and J. G. Patton. 2001. An RNA recognition motif (RRM) is required for the localization of PTB-associated splicing factor (PSF) to subnuclear speckles. Exp Cell Res 263:131-44.
46. Emmott, E., H. Wise, E. M. Loucaides, D. A. Matthews, P. Digard, and J. A. Hiscox. 2010. Quantitative proteomics using SILAC coupled to LC-MS/MS reveals changes in the nucleolar proteome in influenza A virus-infected cells. J Proteome Res 9:5335-45.
47. Eperon, I. C., O. V. Makarova, A. Mayeda, S. H. Munroe, J. F. Caceres, D. G. Hayward, and A. R. Krainer. 2000. Selection of alternative 5' splice sites: role of U1 snRNP and models for the antagonistic effects of SF2/ASF and hnRNP A1. Mol Cell Biol 20:8303-18.
48. Escudero-Paunetto, L., L. Li, F. P. Hernandez, and R. M. Sandri-Goldin. 2010. SR proteins SRp20 and 9G8 contribute to efficient export of herpes simplex virus 1 mRNAs. Virology 401:155-64.
49. Eskiw, C. H., A. Rapp, D. R. Carter, and P. R. Cook. 2008. RNA polymerase II activity is located on the surface of protein-rich transcription factories. J Cell Sci 121:1999-2007.
50. Everett, R. D. 2000. ICP0, a regulator of herpes simplex virus during lytic and latent infection. Bioessays 22:761-70.
51. Everett, R. D. 2006. Interactions between DNA viruses, ND10 and the DNA damage response. Cell Microbiol 8:365-74.
52. Everett, R. D., and M. K. Chelbi-Alix. 2007. PML and PML nuclear bodies: implications in antiviral defence. Biochimie 89:819-30.
53. Everett, R. D., P. Freemont, H. Saitoh, M. Dasso, A. Orr, M. Kathoria, and J. Parkinson. 1998. The disruption of ND10 during herpes simplex virus infection correlates with the Vmw110- and proteasome-dependent loss of several PML isoforms. J Virol 72:6581-91.
54. Fontana, F. 1781. Traité sur le venin de la vipere, avec des observations sur la structure primitive du corps animale. Florence.
55. Fox, A. H., Y. W. Lam, A. K. Leung, C. E. Lyon, J. Andersen, M. Mann, and A. I. Lamond. 2002. Paraspeckles: a novel nuclear domain. Curr Biol 12:13-25.
56. Fox, A. H., and A. I. Lamond. 2011. Paraspeckles. Cold Spring Harb Perspect Biol 2:a000687.
57. Geoffroy, M. C., A. L. Epstein, E. Toublanc, P. Moullier, and A. Salvetti. 2004. Herpes simplex virus type 1 ICP0 protein mediates activation of adeno-associated virus type 2 rep gene expression from a latent integrated form. J Virol 78:10977-86.
58. Geoffroy, M. C., and A. Salvetti. 2005. Helper functions required for wild type and recombinant adeno-associated virus growth. Curr Gene Ther 5:265-71.
59. Greco, A. 2009. Involvement of the nucleolus in replication of human viruses. Rev Med Virol 19:201-14.
60. Grimm, D., and M. A. Kay. 2003. From virus evolution to vector revolution: use of naturally occurring serotypes of adeno-associated virus (AAV) as novel vectors for human gene therapy. Curr Gene Ther 3:281-304.
61. Grinstein, E., P. Wernet, P. J. Snijders, F. Rosl, I. Weinert, W. Jia, R. Kraft, C. Schewe, M. Schwabe, S. Hauptmann, M. Dietel, C. J. Meijer, and H. D. Royer. 2002. Nucleolin as activator of human papillomavirus type 18 oncogene transcription in cervical cancer. J Exp Med 196:1067-78.
62. Hakata, Y., T. Umemoto, S. Matsushita, and H. Shida. 1998. Involvement of human CRM1 (exportin 1) in the export and multimerization of the Rex protein of human T-cell leukemia virus type 1. J Virol 72:6602-7.
63. Hall, L. L., K. P. Smith, M. Byron, and J. B. Lawrence. 2006. Molecular anatomy of a speckle. Anat Rec A Discov Mol Cell Evol Biol 288:664-75.
64. Hamilton, H., J. Gomos, K. I. Berns, and E. Falck-Pedersen. 2004. Adeno-associated virus site-specific integration and AAVS1 disruption. J Virol 78:7874-82.
65. Himmelspach, M., Y. Cavaloc, K. Chebli, J. Stevenin, and R. Gattoni. 1995. Titration of serine/arginine (SR) splicing factors during adenoviral infection modulates E1A pre-mRNA alternative splicing. Rna 1:794-806.
66. Hirano, M., S. Kaneko, T. Yamashita, H. Luo, W. Qin, Y. Shirota, T. Nomura, K. Kobayashi, and S. Murakami. 2003. Direct interaction between nucleolin and hepatitis C virus NS5B. J Biol Chem 278:5109-15.
67. Hozak, P., P. R. Cook, C. Schofer, W. Mosgoller, and F. Wachtler. 1994. Site of transcription of ribosomal RNA and intranucleolar structure in HeLa cells. J Cell Sci 107 ( Pt 2):639-48.
68. Hozak, P., A. B. Hassan, D. A. Jackson, and P. R. Cook. 1993. Visualization of replication factories attached to nucleoskeleton. Cell 73:361-73.
69. Hughes, S. A., H. Wedemeyer, and P. M. Harrison. 2011. Hepatitis delta virus. Lancet 378:73-85.
70. Ibrahim, E. C., T. D. Schaal, K. J. Hertel, R. Reed, and T. Maniatis. 2005. Serine/arginine-rich protein-dependent suppression of exon skipping by exonic splicing enhancers. Proc Natl Acad Sci U S A 102:5002-7.
71. Jablonski, J. A., and M. Caputi. 2009. Role of cellular RNA processing factors in human immunodeficiency virus type 1 mRNA metabolism, replication, and infectivity. J Virol 83:981-92.
72. James, N. J., G. J. Howell, J. H. Walker, and G. E. Blair. 2010. The role of Cajal bodies in the expression of late phase adenovirus proteins. Virology 399:299-311.
73. Jaunin, F., and S. Fakan. 2002. DNA replication and nuclear architecture. J Cell Biochem 85:1-9.
74. Johansson, P. J., E. B. Myhre, and J. Blomberg. 1985. Specificity of Fc receptors induced by herpes simplex virus type 1: comparison of immunoglobulin G from different animal species. J Virol 56:489-94.
75. Kaiser, T. E., R. V. Intine, and M. Dundr. 2008. De novo formation of a subnuclear body. Science 322:1713-7.
76. Koning, R., S. van den Worm, J. R. Plaisier, J. van Duin, J. Pieter Abrahams, and H. Koerten. 2003. Visualization by cryo-electron microscopy of genomic RNA that binds to the protein capsid inside bacteriophage MS2. J Mol Biol 332:415-22.
77. Kotin, R. M. 2011. Large-scale recombinant adeno-associated virus production. Hum Mol Genet 20:R2-6.
78. Kruhlak, M. J., M. A. Lever, W. Fischle, E. Verdin, D. P. Bazett-Jones, and M. J. Hendzel. 2000. Reduced mobility of the alternate splicing factor (ASF) through the nucleoplasm and steady state speckle compartments. J Cell Biol 150:41-51.
79. La Scola, B., C. Desnues, I. Pagnier, C. Robert, L. Barrassi, G. Fournous, M. Merchat, M. Suzan-Monti, P. Forterre, E. Koonin, and D. Raoult. 2008. The virophage as a unique parasite of the giant mimivirus. Nature 455:100-4.
80. Lafarga, M., I. Casafont, R. Bengoechea, O. Tapia, and M. T. Berciano. 2009. Cajal's contribution to the knowledge of the neuronal cell nucleus. Chromosoma 118:437-43.
81. Lallemand-Breitenbach, V., and H. de The. 2010. PML nuclear bodies. Cold Spring Harb Perspect Biol 2:a000661.
82. Lallemand-Breitenbach, V., M. Jeanne, S. Benhenda, R. Nasr, M. Lei, L. Peres, J. Zhou, J. Zhu, B. Raught, and H. de The. 2008. Arsenic degrades PML or PML-RARalpha through a SUMO-triggered RNF4/ubiquitin-mediated pathway. Nat Cell Biol 10:547-55.
83. Lawrence, F. J., B. McStay, and D. A. Matthews. 2006. Nucleolar protein upstream binding factor is sequestered into adenovirus DNA replication centres during infection without affecting RNA polymerase I location or ablating rRNA synthesis. J Cell Sci 119:2621-31.
84. Lee, C. H., S. C. Chang, C. J. Chen, and M. F. Chang. 1998. The nucleolin binding activity of hepatitis delta antigen is associated with nucleolus targeting. J Biol Chem 273:7650-6.
85. Leonard, C. J., and K. I. Berns. 1994. Adeno-associated virus type 2: a latent life cycle. Prog Nucleic Acid Res Mol Biol 48:29-52.
86. Lin, S., G. Coutinho-Mansfield, D. Wang, S. Pandit, and X. D. Fu. 2008. The splicing factor SC35 has an active role in transcriptional elongation. Nat Struct Mol Biol 15:819-26.
87. Linden, R. M., P. Ward, C. Giraud, E. Winocour, and K. I. Berns. 1996. Site-specific integration by adeno-associated virus. Proc Natl Acad Sci U S A 93:11288-94.
88. Lu, H., W. Li, W. S. Noble, D. Payan, and D. C. Anderson. 2004. Riboproteomics of the hepatitis C virus internal ribosomal entry site. J Proteome Res 3:949-57.
89. Mao, Y. S., H. Sunwoo, B. Zhang, and D. L. Spector. 2011. Direct visualization of the co-transcriptional assembly of a nuclear body by noncoding RNAs. Nat Cell Biol 13:95-101.
90. Mao, Y. S., B. Zhang, and D. L. Spector. 2011. Biogenesis and function of nuclear bodies. Trends Genet 27:295-306.
91. Marcus-Sekura, C. J., and B. J. Carter. 1983. Chromatin-like structure of adeno-associated virus DNA in infected cells. J Virol 48:79-87.
92. Matthews, D. A. 2001. Adenovirus protein V induces redistribution of nucleolin and B23 from nucleolus to cytoplasm. J Virol 75:1031-8.
93. Matunis, M. J., X. D. Zhang, and N. A. Ellis. 2006. SUMO: the glue that binds. Dev Cell 11:596-7.
94. McFarlane, M., and S. V. Graham. 2010. Human papillomavirus regulation of SR proteins. Biochem Soc Trans 38:1116-21.
95. McNally, L. M., and M. T. McNally. 1996. SR protein splicing factors interact with the Rous sarcoma virus negative regulator of splicing element. J Virol 70:1163-72.
96. Mingozzi, F., and K. A. High. 2011. Therapeutic in vivo gene transfer for genetic disease using AAV: progress and challenges. Nat Rev Genet 12:341-55.
97. Misteli, T. 2007. Beyond the sequence: cellular organization of genome function. Cell 128:787-800.
98. Misteli, T. 2001. The concept of self-organization in cellular architecture. J Cell Biol 155:181-5.
99. Misteli, T., J. F. Caceres, J. Q. Clement, A. R. Krainer, M. F. Wilkinson, and D. L. Spector. 1998. Serine phosphorylation of SR proteins is required for their recruitment to sites of transcription in vivo. J Cell Biol 143:297-307.
100. Misteli, T., J. F. Caceres, and D. L. Spector. 1997. The dynamics of a pre-mRNA splicing factor in living cells. Nature 387:523-7.
101. Moroy, T., and F. Heyd. 2007. The impact of alternative splicing in vivo: mouse models show the way. Rna 13:1155-71.
102. Muratani, M., D. Gerlich, S. M. Janicki, M. Gebhard, R. Eils, and D. L. Spector. 2002. Metabolic-energy-dependent movement of PML bodies within the mammalian cell nucleus. Nat Cell Biol 4:106-10.
103. Nakai, H., S. R. Yant, T. A. Storm, S. Fuess, L. Meuse, and M. A. Kay. 2001. Extrachromosomal recombinant adeno-associated virus vector genomes are primarily responsible for stable liver transduction in vivo. J Virol 75:6969-76.
104. Nash, K., W. Chen, W. F. McDonald, X. Zhou, and N. Muzyczka. 2007. Purification of host cell enzymes involved in adeno-associated virus DNA replication. J Virol 81:5777-87.
105. Nash, K., W. Chen, and N. Muzyczka. 2008. Complete in vitro reconstitution of adeno-associated virus DNA replication requires the minichromosome maintenance complex proteins. J Virol 82:1458-64.
106. Nash, K., W. Chen, M. Salganik, and N. Muzyczka. 2009. Identification of cellular proteins that interact with the adeno-associated virus rep protein. J Virol 83:454-69.
107. Nasr, R., E. Chiari, M. El-Sabban, R. Mahieux, Y. Kfoury, M. Abdulhay, V. Yazbeck, O. Hermine, H. de The, C. Pique, and A. Bazarbachi. 2006. Tax ubiquitylation and sumoylation control critical cytoplasmic and nuclear steps of NF-kappaB activation. Blood 107:4021-9.
108. Ni, T. H., W. F. McDonald, I. Zolotukhin, T. Melendy, S. Waga, B. Stillman, and N. Muzyczka. 1998. Cellular proteins required for adeno-associated virus DNA replication in the absence of adenovirus coinfection. J Virol 72:2777-87.
109. Nicolas, A., N. Alazard-Dany, C. Biollay, L. Arata, N. Jolinon, L. Kuhn, M. Ferro, S. K. Weller, A. L. Epstein, A. Salvetti, and A. Greco. 2010. Identification of rep-associated factors in herpes simplex virus type 1-induced adeno-associated virus type 2 replication compartments. J Virol 84:8871-87.
110. Nieto, K., C. Stahl-Hennig, B. Leuchs, M. Muller, L. Gissmann, and J. A. Kleinschmidt. 2012. Intranasal Vaccination with AAV5 and 9 Vectors Against Human Papillomavirus Type 16 in Rhesus Macaques. Hum Gene Ther.
111. Ote, I., M. Lebrun, P. Vandevenne, S. Bontems, C. Medina-Palazon, E. Manet, J. Piette, and C. Sadzot-Delvaux. 2009. Varicella-zoster virus IE4 protein interacts with SR proteins and exports mRNAs through the TAP/NXF1 pathway. PLoS One 4:e7882.
112. Penaud-Budloo, M., C. Le Guiner, A. Nowrouzi, A. Toromanoff, Y. Cherel, P. Chenuaud, M. Schmidt, C. von Kalle, F. Rolling, P. Moullier, and R. O. Snyder. 2008. Adeno-associated virus vector genomes persist as episomal chromatin in primate muscle. J Virol 82:7875-85.
113. Perry, R. P. 1962. The Cellular Sites of Synthesis of Ribosomal and 4s Rna. Proc Natl Acad Sci U S A 48:2179-86.
114. Phair, R. D., and T. Misteli. 2000. High mobility of proteins in the mammalian cell nucleus. Nature 404:604-9.
115. Qiu, J., and K. E. Brown. 1999. A 110-kDa nuclear shuttle protein, nucleolin, specifically binds to adeno-associated virus type 2 (AAV-2) capsid. Virology 257:373-82.
116. Qiu, J., and D. J. Pintel. 2002. The adeno-associated virus type 2 Rep protein regulates RNA processing via interaction with the transcription template. Mol Cell Biol 22:3639-52.
117. Rabl, C. 1885. Über Zelltheilung. Morph Jb 10:214-330.
118. Raska, I., L. E. Andrade, R. L. Ochs, E. K. Chan, C. M. Chang, G. Roos, and E. M. Tan. 1991. Immunological and ultrastructural studies of the nuclear coiled body with autoimmune antibodies. Exp Cell Res 195:27-37.
119. Rosa-Calatrava, M., F. Puvion-Dutilleul, P. Lutz, D. Dreyer, H. de The, B. Chatton, and C. Kedinger. 2003. Adenovirus protein IX sequesters host-cell promyelocytic leukaemia protein and contributes to efficient viral proliferation. EMBO Rep 4:969-75.
120. Roscigno, R. F., and M. A. Garcia-Blanco. 1995. SR proteins escort the U4/U6.U5 tri-snRNP to the spliceosome. Rna 1:692-706.
121. Ruggero, D., and P. P. Pandolfi. 2003. Does the ribosome translate cancer? Nat Rev Cancer 3:179-92.
122. Said, E. A., J. Courty, J. Svab, J. Delbe, B. Krust, and A. G. Hovanessian. 2005. Pleiotrophin inhibits HIV infection by binding the cell surface-expressed nucleolin. Febs J 272:4646-59.
123. Saitoh, N., C. S. Spahr, S. D. Patterson, P. Bubulya, A. F. Neuwald, and D. L. Spector. 2004. Proteomic analysis of interchromatin granule clusters. Mol Biol Cell 15:3876-90.
124. Sasaki, Y. T., T. Ideue, M. Sano, T. Mituyama, and T. Hirose. 2009. MENepsilon/beta noncoding RNAs are essential for structural integrity of nuclear paraspeckles. Proc Natl Acad Sci U S A 106:2525-30.
125. Schneider, J., B. Dauber, K. Melen, I. Julkunen, and T. Wolff. 2009. Analysis of influenza B Virus NS1 protein trafficking reveals a novel interaction with nuclear speckle domains. J Virol 83:701-11.
126. Schnepp, B. C., R. L. Jensen, C. L. Chen, P. R. Johnson, and K. R. Clark. 2005. Characterization of adeno-associated virus genomes isolated from human tissues. J Virol 79:14793-803.
127. Semmes, O. J., and K. T. Jeang. 1996. Localization of human T-cell leukemia virus type 1 tax to subnuclear compartments that overlap with interchromatin speckles. J Virol 70:6347-57.
128. Sharma, A., H. Takata, K. Shibahara, A. Bubulya, and P. A. Bubulya. 2010. Son is essential for nuclear speckle organization and cell cycle progression. Mol Biol Cell 21:650-63.
129. Shen, H., J. L. Kan, and M. R. Green. 2004. Arginine-serine-rich domains bound at splicing enhancers contact the branchpoint to promote prespliceosome assembly. Mol Cell 13:367-76.
130. Shevtsov, S. P., and M. Dundr. 2011. Nucleation of nuclear bodies by RNA. Nat Cell Biol 13:167-73.
131. Shopland, L. S., C. V. Johnson, M. Byron, J. McNeil, and J. B. Lawrence. 2003. Clustering of multiple specific genes and gene-rich R-bands around SC-35 domains: evidence for local euchromatic neighborhoods. J Cell Biol 162:981-90.
132. Sirri, V., P. Roussel, and D. Hernandez-Verdun. 1999. The mitotically phosphorylated form of the transcription termination factor TTF-1 is associated with the repressed rDNA transcription machinery. J Cell Sci 112 ( Pt 19):3259-68.
133. Sivachandran, N., J. Y. Cao, and L. Frappier. 2010. Epstein-Barr virus nuclear antigen 1 Hijacks the host kinase CK2 to disrupt PML nuclear bodies. J Virol 84:11113-23.
134. Sleeman, J. E., and A. I. Lamond. 1999. Newly assembled snRNPs associate with coiled bodies before speckles, suggesting a nuclear snRNP maturation pathway. Curr Biol 9:1065-74.
135. Snyder, R. O., D. S. Im, and N. Muzyczka. 1990. Evidence for covalent attachment of the adeno-associated virus (AAV) rep protein to the ends of the AAV genome. J Virol 64:6204-13.
136. Solimando, L., M. S. Luijsterburg, L. Vecchio, W. Vermeulen, R. van Driel, and S. Fakan. 2009. Spatial organization of nucleotide excision repair proteins after UV-induced DNA damage in the human cell nucleus. J Cell Sci 122:83-91.
137. Sonntag, F., K. Schmidt, and J. A. Kleinschmidt. 2010. A viral assembly factor promotes AAV2 capsid formation in the nucleolus. Proc Natl Acad Sci U S A 107:10220-5.
138. Spector, D. L. 2001. Nuclear domains. J Cell Sci 114:2891-3.
139. Spector, D. L. 1993. Nuclear organization of pre-mRNA processing. Curr Opin Cell Biol 5:442-7.
140. Spector, D. L., and A. I. Lamond. 2010. Nuclear speckles. Cold Spring Harb Perspect Biol 3.
141. Stanek, D., S. D. Rader, M. Klingauf, and K. M. Neugebauer. 2003. Targeting of U4/U6 small nuclear RNP assembly factor SART3/p110 to Cajal bodies. J Cell Biol 160:505-16.
142. Stracker, T. H., G. D. Cassell, P. Ward, Y. M. Loo, B. van Breukelen, S. D. Carrington-Lawrence, R. K. Hamatake, P. C. van der Vliet, S. K. Weller, T. Melendy, and M. D. Weitzman. 2004. The Rep protein of adeno-associated virus type 2 interacts with single-stranded DNA-binding proteins that enhance viral replication. J Virol 78:441-53.
143. Swift, H. 1959. Studies on nuclear fine structure. Brookhaven Symp Biol 12:134-52.
144. Swindle, C. S., N. Zou, B. A. Van Tine, G. M. Shaw, J. A. Engler, and L. T. Chow. 1999. Human papillomavirus DNA replication compartments in a transient DNA replication system. J Virol 73:1001-9.
145. Verma, D., S. Bais, M. Gaillard, and S. Swaminathan. 2010. Epstein-Barr Virus SM protein utilizes cellular splicing factor SRp20 to mediate alternative splicing. J Virol 84:11781-9.
146. Vincent-Lacaze, N., R. O. Snyder, R. Gluzman, D. Bohl, C. Lagarde, and O. Danos. 1999. Structure of adeno-associated virus vector DNA following transduction of the skeletal muscle. J Virol 73:1949-55.
147. Walker, M. P., L. Tian, and A. G. Matera. 2009. Reduced viability, fertility and fecundity in mice lacking the cajal body marker protein, coilin. PLoS One 4:e6171.
148. Walz, C., A. Deprez, T. Dupressoir, M. Durst, M. Rabreau, and J. R. Schlehofer. 1997. Interaction of human papillomavirus type 16 and adeno-associated virus type 2 co-infecting human cervical epithelium. J Gen Virol 78 ( Pt 6):1441-52.
149. Wang, J., J. Xie, H. Lu, L. Chen, B. Hauck, R. J. Samulski, and W. Xiao. 2007. Existence of transient functional double-stranded DNA intermediates during recombinant AAV transduction. Proc Natl Acad Sci U S A 104:13104-9.
150. Wansink, D. G., W. Schul, I. van der Kraan, B. van Steensel, R. van Driel, and L. de Jong. 1993. Fluorescent labeling of nascent RNA reveals transcription by RNA polymerase II in domains scattered throughout the nucleus. J Cell Biol 122:283-93.
151. Weindler, F. W., and R. Heilbronn. 1991. A subset of herpes simplex virus replication genes provides helper functions for productive adeno-associated virus replication. J Virol 65:2476-83.
152. Wistuba, A., A. Kern, S. Weger, D. Grimm, and J. A. Kleinschmidt. 1997. Subcellular compartmentalization of adeno-associated virus type 2 assembly. J Virol 71:1341-52.
153. Wu, Z., A. Asokan, and R. J. Samulski. 2006. Adeno-associated virus serotypes: vector toolkit for human gene therapy. Mol Ther 14:316-27.
154. Xiao, R., Y. Sun, J. H. Ding, S. Lin, D. W. Rose, M. G. Rosenfeld, X. D. Fu, and X. Li. 2007. Splicing regulator SC35 is essential for genomic stability and cell proliferation during mammalian organogenesis. Mol Cell Biol 27:5393-402.
155. Xie, Q., W. Bu, S. Bhatia, J. Hare, T. Somasundaram, A. Azzi, and M. S. Chapman. 2002. The atomic structure of adeno-associated virus (AAV-2), a vector for human gene therapy. Proc Natl Acad Sci U S A 99:10405-10.
156. Xu, H., R. S. Pillai, T. N. Azzouz, K. B. Shpargel, C. Kambach, M. D. Hebert, D. Schumperli, and A. G. Matera. 2005. The C-terminal domain of coilin interacts with Sm proteins and U snRNPs. Chromosoma 114:155-66.
157. Zhang, H. G., Y. M. Wang, J. F. Xie, X. Liang, H. C. Hsu, X. Zhang, J. Douglas, D. T. Curiel, and J. D. Mountz. 2001. Recombinant adenovirus expressing adeno-associated virus cap and rep proteins supports production of high-titer recombinant adeno-associated virus. Gene Ther 8:704-12.
158. Zhong, L., X. Zhou, Y. Li, K. Qing, X. Xiao, R. J. Samulski, and A. Srivastava. 2008. Single-polarity recombinant adeno-associated virus 2 vector-mediated transgene expression in vitro and in vivo: mechanism of transduction. Mol Ther 16:290-5.
159. Zhu, Y., R. L. Tomlinson, A. A. Lukowiak, R. M. Terns, and M. P. Terns. 2004. Telomerase RNA accumulates in Cajal bodies in human cancer cells. Mol Biol Cell 15:81-90.
